微生物群落重要而复杂,而铁的博弈可能是理解微生物博弈的一个重要切入点。过去的两年中,我们课题组和合作者发表了多篇以定量角度切入微生物铁互作的工作。2025年1月10日,综合之前的工作和思考未来的方向,我们在Quantitative Biology上刊发了一篇观点文章:“Forging the iron-net: Towards a quantitative understanding of microbial communities via siderophore-mediated interactions” (doi.org/10.1002/qub2.84),总结了我们与合作者最近关于微生物铁互作的一系列工作所指向的新方向——以计算方法解析微生物的铁互作网络——这是一个刚刚起步的、有趣而充满挑战性的领域,我们希望有更多朋友加入这个有趣的探索之旅。
目录
构筑铁网:定量刻画微生物王国中的铁资源版图
1.一张连接微生物群落的庞大铁网络
缺铁悖论:细菌创造的“贫铁时代”
铁载体和受体:多样的“铁专属快递”
特异性配对:铁载体系统的“钥匙和锁”
借机取巧:细菌间的“顺手牵羊”
2. 从序列到网络——数据时代的“抄近路”
假设1:自己造的铁载体自己得吃
共进化的密码:基因里的“钥匙-锁”配对印记
假设2:序列相似性~功能相似性
下一步,走向更大的“铁网”
3. “野餐聚会”——铁博弈中的游戏规则
三类铁行为:老实、滑头、纯蹭饭
病原菌——POTLUCK聚会中的“不被欢迎者”?
微生物互作:博弈论的游乐场
4. “相邻可能性”——永不停歇的化学创新
研究者团队
计算团队:北京大学前沿交叉学科研究院-李志远团队
实验团队:南京农业大学LorMe韦中团队
相关文章
1.一张连接微生物群落的庞大铁网络
缺铁悖论:细菌创造的“贫铁时代”
说来离谱,这个地球上,几乎所有生物都缺铁。铁是恒星核聚变的终点产物,在宇宙中分布广泛,更是占据了地球总质量的1/3以上。按理说,我们就不该听说“缺铁”这回事[1]。
铁在生物中又是个很好用的元素。这很大程度上要归功于它独特的化学性质——它能在二价铁(Fe²⁺)和三价铁(Fe³⁺)之间自如转换,提供恰到好处的氧化还原电位,并能形成多种配位结构。这些特性使得铁成为电子传递、发酵和呼吸作用、DNA合成与修复、以及多种重要酶促反应中的关键中心。。
在原始的海洋中,溶解态的铁曾是应有尽有的——地壳中第四丰富的元素呢。然而情况在大约24亿年前发生了戏剧性的转变。罪魁祸首是一群叫做蓝细菌(也称蓝藻)的微生物,它们在进行光合作用时,不断地将二氧化碳转化为氧气。经过漫长的时间积累,地球的大气层逐渐被氧气充满,这一事件被称为“大氧化事件”。氧气的大量存在导致可溶性的二价铁迅速被氧化成三价铁,而三价铁的溶解度极低,在中性pH环境下几乎全部以不溶性的氧化物形式存在。这下,细菌傻眼了(图 1)。
由此看来,缺铁这件事几乎是细菌的“自食其果”——从进化生态学的角度,这是一个绝妙的案例,展示了适应度地形(fitness landscape)并非一成不变,而是会被生物的行为所重塑[2]。蓝细菌以一己之力将有氧呼吸拱上了适应度地形图的高点,也将整个地球生物圈拖入了极度缺铁的状态。
面对这一困境,古老年代的微生物们似乎有两个选择:要么放弃铁,转而寻找其他金属元素来完成催化和电子传递的功能;要么想办法更有效地获取环境中的铁。那么,微生物们最终选择了哪条路呢?
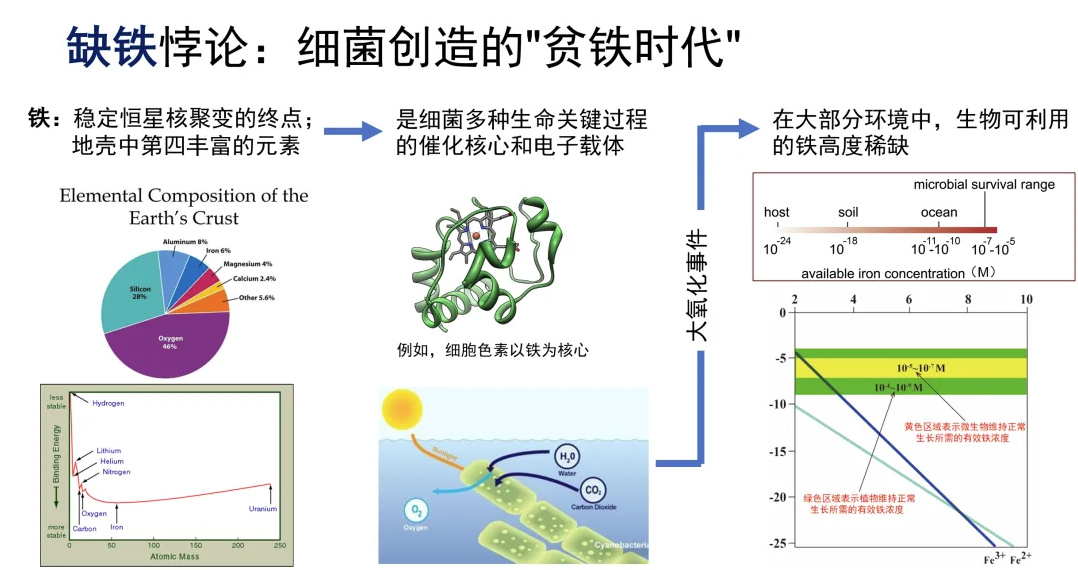
图1. 在蓝细菌所诱导的大氧化事件后,氧化后的三价铁浓度极低,整个地球进入“贫铁时代”
铁载体和受体:多样的“铁专属快递”
两条路都有菌去选。有一些“壮士断腕”的微生物选择了第一条路。比如支原体,这种号称“极简主义者”的微生物,通过删除大量依赖铁的代谢系统,将对铁的需求降到了最低;而一些乳酸菌则巧妙地用锰来替代部分铁的功能。但有趣的是,即便是这些“另类”,也没有完全放弃使用铁——铁的某些功能实在是难以完全被替代。
事实上,写在基因组中的历史告诉我们,“想办法搞铁”是大多数微生物的选择,而大部分微生物搞铁的方式是通过一类叫做铁载体(siderophore)的小分子分泌物——这件略显离谱的事,还是19年时南农的韦老师告诉我的,确实每次跟真正的搞生物的吃饭都能学到点新东西——这篇文中的大部分工作,都是我们两个团队理论配合实验一起做的(前情提要)。
铁载体是一类能够强力螯合铁离子的小分子,就像是微生物们专门设计的“铁离子夹子”。它们被分泌到细胞外,在环境中“巡逻”,一旦遇到铁离子就紧紧抓住(图 2)。
目前研究者已经发现了超过700种不同的铁载体结构,它们的相关信息都被收录在我们于2023年上线的SIDERITE数据库中[3](铁载体信息数据库SIDERITE与天然产物化学空间中暗藏的多样性)。我们对数据库中的结构进行了详细的分析——功能基团、来源、分子量……这些迥异的结构都有一个共同点:具有能螯合铁的基团(图 2)。更有意思的是,当我们根据这些特征,去更大的天然产物数据库中探索时,发现还有数以千计的分子可能具有抓取铁离子的能力,只是它们还从未被认定为铁载体——已知的700多种铁载体结构,很可能只是这个庞大家族中的一小部分。

图2. 微生物通过铁载体摄取铁的机制:首先,生产和分泌铁载体到环境中;铁载体螯合铁形成复合物,复合物和细胞膜表面的识别蛋白所结合并转运入细胞
铁载体是怎么来的呢?制造这些铁载体的“生产线”主要有两种:一种是“高级定制”路线——非核糖体肽合成酶(NRPS)通路。这条生产线就像是一个精密的工厂,由多个“车间”(也就是功能模块)首尾相连,每个车间都能在分子上安装特定的零件。通过不同车间的组合,这条生产线能够制造出结构复杂、功能强大的铁载体。目前已知的铁载体中,约70%都出自这条生产线。实际上,NRPS这类生产线不仅生产铁载体,还能制造抗生素、毒素等多种帮助微生物适应环境的重要分子,它的演化和功能也是我们课题组的重点研究方向[4](标准化框架促进NRPS的功能预测和结构分析)。另一种是“简约”路线——不依赖NRPS的铁载体合成酶(NIS)通路,虽然产品结构相对简单,但同样能完成任务。
但是,仅仅把铁抓住还不够,还得把它安全送回细胞内。为此,微生物们还进化出了专门的“收货系统”。细菌界的两大阵营在这方面各有妙招:两层膜的革兰氏阴性菌(如大肠杆菌)使用一种叫做TonB依赖型受体的跨膜蛋白,就像是在细胞外膜表面装了一个特制的“货运通道”;而单层膜的革兰氏阳性菌(如枯草芽孢杆菌)则采用周质结合蛋白,像是在细胞壁外布置了一群“抓取工”(图 2)。
当我们对铁载体的合成基因和受体有了深入了解,就可以从基因组序列中对它们进行直接推测。在我们2024年正式发表于eLife上的工作中[5](基因组数据挖掘揭示细菌铁载体通路的隐藏多样性),我们在假单胞这个细菌属中发展了一种“尽量忽略演化干扰”的基因组注释算法,可以从基因组序列中准确预测它们会产生什么样的铁载体(假单胞菌中的典型铁载体是NRPS途径合成的pyoverdine),还可以注释出此前找不到的pyoverdine受体的位置。我们发现:隐藏于基因组中的铁载体多样性远超此前所知——已知假单胞菌属中有37种pyoverdine结构,现在看来至少有188种;已知3种受体亚类,我们预测出大概90种……
这种惊人的多样性提出了一个有趣的问题:在生物进化中,重要的东西往往高度保守,很少发生变化。但铁载体却呈现出如此丰富的多样性,这是否意味着它们不那么重要呢?可是,我们发现超过60%的微生物都保留着制造铁载体的基因,这个比例远高于其它次级代谢产物(比如抗生素,只有1-10%的微生物携带其合成基因)——细菌不养闲基因,那么,为什么微生物需要如此多样的铁载体呢?
特异性配对:铁载体系统的“钥匙和锁”
在前人对铜绿假单胞菌这个物种的研究中,科学家们发现了另一个有趣的现象:不同株系的铜绿假单胞菌虽然都能制造类似的铁载体(pyoverdine),但它们之间却不能通用。就像是每个株系都有自己特制的“钥匙”(铁载体)和“锁”(受体)一样(图 3)。
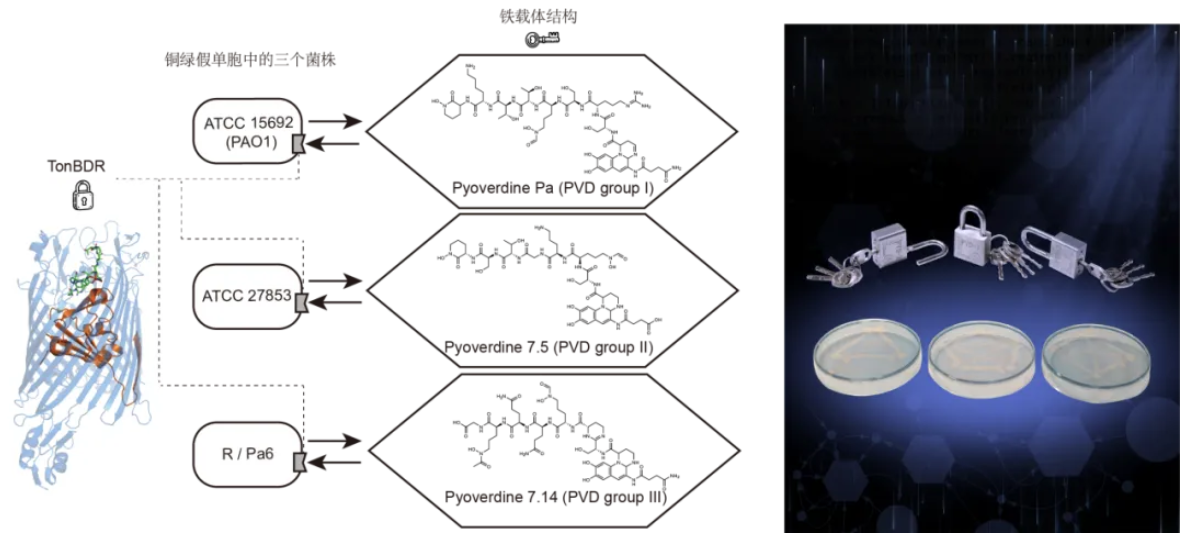
图3. 铁载体和受体之间高度特异的配对关系。图左为三种不同菌株各自产生的铁载体(图中六边形内的分子结构)和它们专属的受体蛋白(灰色凹形标记);图右为竞争封面(未成功)的对这一“锁-钥匙”特异性关系的形象诠释
比如说,同样是铜绿假单胞菌这一个物种,PAO1、ATCC27853和Pa6这三个菌株,每个都制造自己独特的pyoverdine类型(如图 3所示,分别被命名为I型、II型和III型)。更有意思的是,每个菌株只能识别和使用自己制造的那一型pyoverdine,对其他型号却“视而不见”。这种高度的特异性就像是一个精心设计的安保系统——每把钥匙都只能打开与之配套的锁。研究者们通过结构研究推测,这种特异性主要取决于受体蛋白(也就是“锁”)上的某些特定区域。就像是锁芯中的凹凸结构决定了钥匙的形状,这些蛋白质结构决定了哪种铁载体可以被识别和运输。
既然铁载体和受体之间的特异性这么高,那么,是不是每个菌株就造自己的铁载体吃自己的铁载体,跟“别人”没什么关系了呢?
借机取巧:细菌间的“顺手牵羊”
当然不是。微生物可奸诈了。
让我们看看大肠杆菌(E. coli)这个老油条(图 4A):它不仅能吃自己生产的肠杆菌素(enterobactin——这是已知的最强悍的铁载体),还在基因组中保有多种能摄取别的铁载体的受体(图中彩色旗形标记),用来使用其他微生物产生的铁载体——比如克雷伯氏菌产的aerobactin、革兰氏阳性的红球菌产的heterobactin A,甚至真菌产的coprogen也能来一口。
枯草芽孢杆菌(B. subtilis)也是个借东西的高手。它虽然自己只生产芽孢杆菌素(bacillibactin),但却也装备了多个受体,可以使用包括肠杆菌素、铁氧胺E(ferrioxamine E)等在内的多种别人家的铁载体(图 4A)。
铜绿假单胞菌(P. aeruginosa)就更有意思了:它自己产的铁载体(pyoverdines)似乎仅限于假单胞菌这个属内,但基因组中往往有对应着肠杆菌素和铁载菌素(ferrichrome)的受体基因(图 4A)。
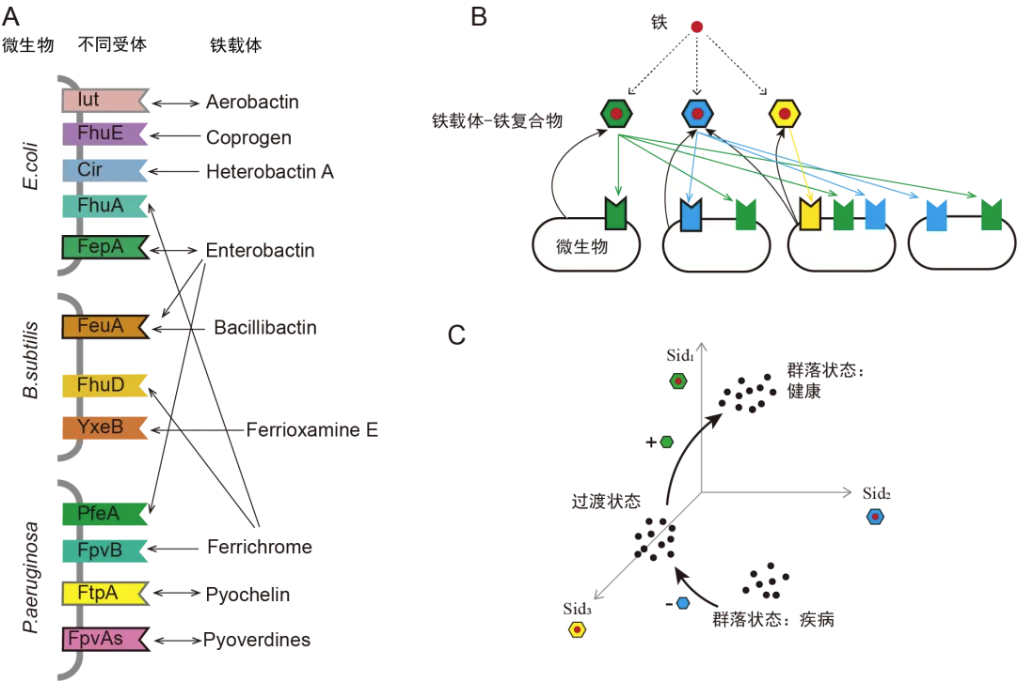
图4. 铁载体网络。A.细菌间铁载体的“盗用”行为:不同细菌(大肠杆菌、枯草芽孢杆菌和铜绿假单胞菌)利用不同的受体(彩色旗形标记)识别和使用不同的铁载体。双向箭头表示自产自用,单向箭头表示盗用它人产生的铁载体。B. 铁网络的概念示意图:微生物(底部圆角方框)通过产生和识别不同类型的铁载体(六边形,不同颜色代表不同种类的铁载体,会被对应颜色的受体吸收),形成复杂的互动网络。C.通过调控不同类型的铁载体,有望实现对微生物群落状态的定向调控。
这些例子反映了一个更普遍的规律:我们还未发表的初步分析发现,虽然只有约60%的微生物有铁载体合成簇,但超过90%的微生物都装备了至少一个铁载体受体基因。这种巨大的差异暗示着在微生物世界中,顺手牵羊的“盗用”比“生产”或许更常见。
这些发现揭示了一张庞大的“铁网”:铁载体的多样性、铁载体-受体间的特异性配对关系,以及普遍存在的“盗用”现象,将微生物编织成了一个错综复杂的铁素相互作用网络——每个物种既可能是某种铁载体的生产者,也可能是其他铁载体的盗用者。这张网络之所以特别,在于它既有普遍性——因为铁对所有微生物都必不可少;又有特异性——因为不同的铁载体系统可以选择性地被不同微生物使用。一旦我们能弄清楚每个微生物制造什么、借用什么样的铁载体,我们就能以绘出这张“铁网”(图 4B)。
微生物群落重要却复杂,难以被调控——给抗生素一起死给营养一起活。而铁载体这种能精确到菌株水平的差异性,或许能成为调控微生物群落的一个有利“抓手”(图 4C)。理想总是美好的。
现实总是艰难的——精确到菌株水平的差异性也带来了让人头秃的复杂性。光是铜绿假单胞菌这一个物种中的三个菌株就有三种不同的铁载体,而整个微生物世界可是有着3000多个属(已知)。如果要用实验去测试它们之间的两两互作关系,学生和我至少得崩一个。
那么,在这个数据爆炸的时代,我们有什么“抄近路”的办法吗?
2. 从序列到网络——数据时代的“抄近路”
我们2025年1月发在Science Advances上的这篇文章,在假单胞菌这个属中迈出了铁载体领域“从序列到网络”的第一步[6](基于基因组数据挖掘高效预测微生物铁载体互作网络)。简单说,我们找到了一条“近路”:通过分析大量基因组中的共进化信息,推断出每个菌株制造什么样的铁载体、吸收什么样的铁载体,从而重构出整个铁相互作用网络。
这个方法基于两个关键假设和它们带来的推论——做理论的都知道假设能多离谱,但咱们先这么假设着试试看:
假设1:自己造的铁载体自己得吃
这个假设朴素但还算合理——因为制造铁载体的成本实在太高了。NRPS是微生物中最大的酶之一,还跟核糖体竞争底物。前人有实验表明,pyoverdine的合成大概会把生长速度拖慢5%左右——这个劣势对繁殖速度以小时为量级微生物来说已经几乎难以承受了。这么高的代价下,如果连自己产的铁载体都用不了,那不是白白浪费资源吗?所以,无论一个微生物顺了多少别人的铁载体,我们假设,只要它生产铁载体,它就至少得有一个能识别自己产的铁载体的受体。
共进化的密码:基因里的“钥匙-锁”配对印记
假设1带来了一个重要推论:铁载体合成基因和它对应的受体基因之间必然存在共进化关系。就像图 5A所展示的,如果合成基因发生了变化,对应的受体基因就必须跟着改变,否则这对“钥匙和锁”就对不上了;反之亦然:如果受体基因发生了突变,合成基因也得跟着变。这种共同进化的信号,就像是留在基因中的印记,让我们能够将正确的“钥匙”和“锁”配对起来。
由此,我们发展了一套不是很深度的算法,在我们之前注释好的1928个假单胞菌基因组中,以最大化共进化强度的方法,把哪些合成基因和哪些受体基因形成“锁钥对”给找出来了——我们找到了49个这样的“配对”(图 5B)。
PS1:共进化方法在生物信息学中应用广泛。比如在结构预测领域,包括AlphaFold在内的许多方法都利用了蛋白质序列中的共进化信息来预测氨基酸之间的空间接触。
PS2: NRPS天生就容易变——这也使它成为微生物军备竞赛中的重要武器[7, 8](【作者有话说】夏威夷寄生菌,演化中的“海浪”,和“还有这事”?)
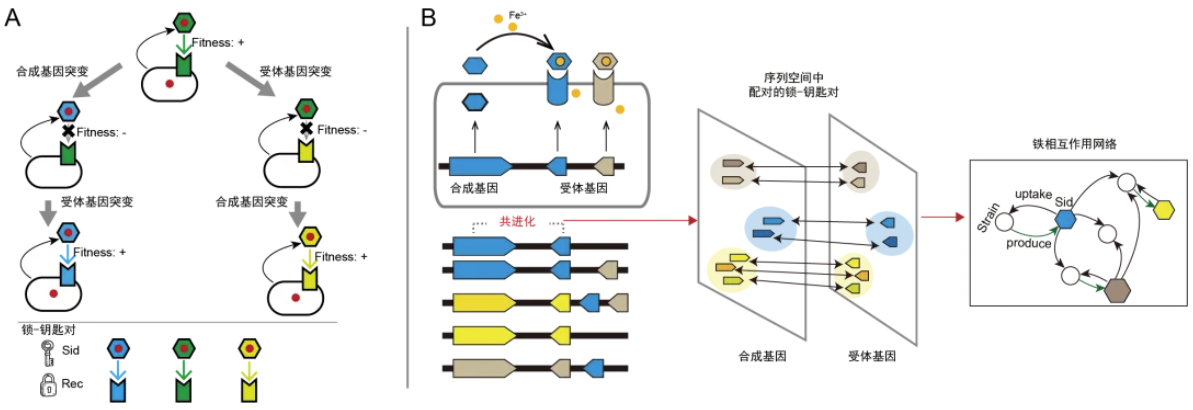
图5. 从基因到网络的推断原理。A. 共进化的驱动力:铁载体(六边形)和其受体(旗形)必须配对(颜色一致)才能发挥功能。当一方的结构发生变化时(以颜色变化表示),另一方必须随之改变以维持功能,否则细菌的生存能力(fitness)会下降。B. 从基因序列重建铁网络的方法:通过分析基因序列中的共进化信号,我们可以推断铁载体合成基因与受体基因的配对关系,进而重建整个铁互作网络。
假设2:序列相似性~功能相似性
有了“锁钥对”后,接下的关键问题是:当我们看到一个菌株有多个铁载体受体时,除了能识别自己产的铁载体的那个“自受体”,其他“盗受体”究竟在偷谁家的铁载体?如果假设2成立,那么答案就简单了:看这些受体基因的序列跟哪个已知的“锁钥对”更相似,就能推测出它们可能在使用谁家的铁载体。
PS: “序列相似性等同于功能相似性”这个假设在演化距离不太远,比如还在一个科或属之内时,基本成立,但一旦演化距离大了,微生物就有自己的想法了,这个假设也许就要出问题了。
通过这两个假设所带来的计算模型,我们就能确定每个菌株的“铁行为特征”——它造哪种类型,吃哪种类型的铁载体。就像是给每个菌株都贴上了一个“铁行为标签”。据这些标签,我们就将所有菌株连接成一张庞大的互动网络(图 5B,最后一个图)。
LorMe实验室为这些预测做了实验,准确率在90%以上,嘿。
关于这篇工作的更多细节,可以在【基于基因组数据挖掘高效预测微生物铁载体互作网络】里看到。
下一步,走向更大的“铁网”
假单胞属中的成功只是一个开始。图 6A展示了我们接下来去配对更多“锁-钥配对”的思路:例如基因的位置关系(在相当一部分菌里不好用)、序列共进化(我们在假单胞属中用的)、以及通过分子对接预测相互作用(未来可期)。
但是,整个微生物世界实在太大了。我们这次的发表只是在铁网的拼图里填上了小小一块。放眼望去,还有数千个细菌属、数百个真菌属等待探索,而且接下来还得把不同种属统一起来。更何况,铁载体系统只是微生物世界众多互动系统中的一个。抗生素、毒素、群体感应……这些次级代谢产物都涉及多个蛋白之间的互动,都可能留下共进化的印记。
事实上,当我们试图跨越更远的物种界限时,序列分析方法已经显露出力不从心。这时候,我们选择了“打不过人工智能就加入”的策略——如图 6B所示,我们期望通过人工智能的方法,最终建立一个通用的预训练模型,让它能从任意微生物的基因组中直接预测出这个微生物的“铁行为特征”
这个愿景野心勃勃,但也充满希望。毕竟,每一次技术的进步都在帮助我们更好地理解这个微观世界的社交网络——这也将是一个有趣、庞大、且复杂,需要理论和实验一起来投入的巨大拼图,我们期待更多朋友能加入这场充满挑战与乐趣的拼图游戏。
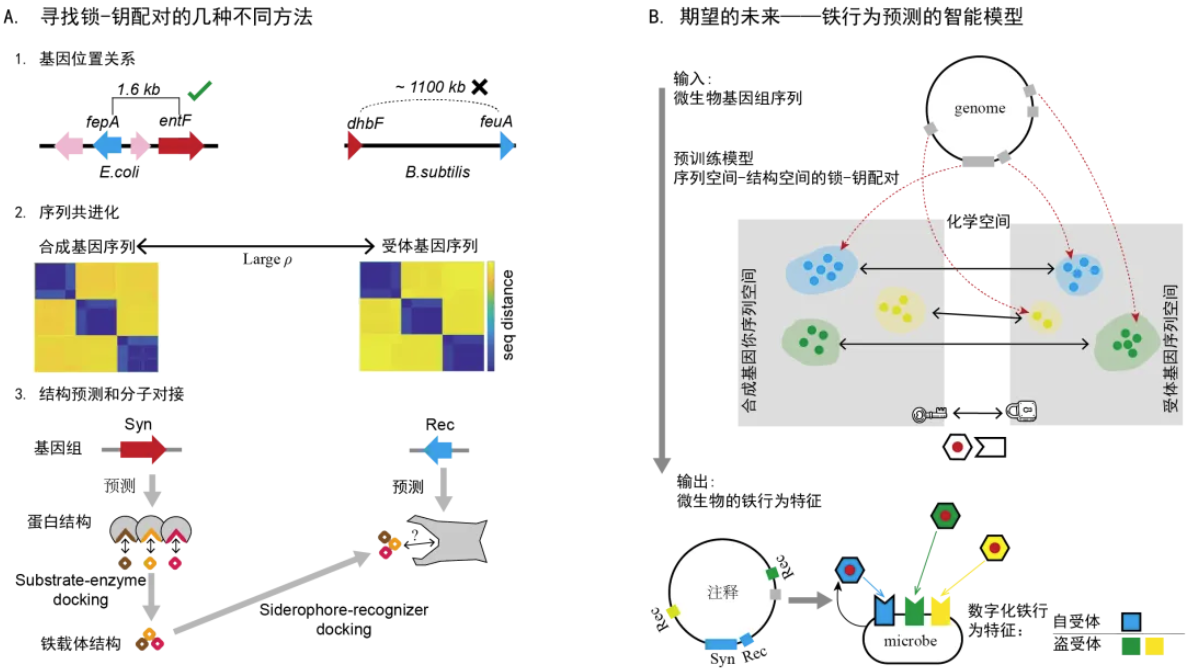
图6. 探索铁网络的过去与未来。A. 寻找铁载体-受体配对关系的三种方法:通过基因位置关系(上)、序列共进化分析(中)、以及分子结构预测(下) B. 面向未来的智能预测:我们期望建立一个预训练模型,能够直接从基因组序列预测微生物的铁行为特征。
3. “野餐聚会”——铁博弈中的游戏规则
三类铁行为:老实、滑头、纯蹭饭
我这里邀请大家加入这个铁网络的拼图游戏,是因为这个网络目测还挺好玩的——比如,光是数数每个基因组中有几个铁载体的合成基因和受体基因,就能看见三大类不同的“行为”(图 7A):
1. 学名“单受体生产者”,别名“老实人”——只生产一种铁载体,也只有对应的那一个受体,不占别人的便宜。
2. 学名“多受体生产者”,别名“小滑头”——既生产自己的铁载体,又装备了多个不同的受体——组会里我们也叫它“鸡贼菌”——既能占别人的便宜,也保证只有自己存在于环境中时有独立摄取铁的能力。
3. 学名“非生产者”,别名“纯蹭饭的”——基因组里压根就没有铁载体合成基因,但却有着一个或多个受体。虽然看起来有点不地道,但这种策略在自然界中也不算少——毕竟,如果可以轻松吃现成的,为什么要辛辛苦苦做菜呢?
多受体生产者“小滑头”连接起了不同种类的铁载体,将这将近2000个菌株连成一张巨大的互作网络(图 7B)。这个网络复杂而美丽,长在那里跟朵花儿似的——我们还能从它里面看出什么?

图7. 三类铁行为特征(A)和假单胞菌属的铁互作网络(B)
病原菌——POTLUCK聚会中的“不被欢迎者”?
对于做计算的人,大多数在写代码的时候就知道程序会给你吐什么结果了。什么时候最好玩?——结果让你惊一下的时候(& without bug)。
那是一年前被某期刊编辑说not of broad interest被拒后的百无聊赖,让我点着上面那个巨大的属水平的网络,一个一个物种提出来看个好玩。
先看菌株数最多的铜绿假单胞菌——臭名昭著的人体致病菌,以多重耐药而闻名。这铁网络咋这么简单?几乎就三组铁载体-受体配对,三大类老实菌各自安好(图 8,第一行第一个)。
菌株第二多的是土壤菌荧光假单胞菌,等等这铁网络怎么这么复杂,是土壤菌比较自由散漫么?(图 8,第二行第一个)
第三多的是个植物病原菌,你平时也在土壤里对吧,怎么突然又简单回去了?(图 8,第一行第二个)。
继续一个物种一个物种看下去,一个有趣的模式开始浮现:非病原菌的网络则复杂得多,且大多表现出“滑头”(黄色)的特征。然而,不管是感染动物还是植物的病原菌,它们的铁网络却都出奇的简单,且要么是特别“老实”(绿色),要么是纯粹“蹭饭”(红色);此外,病原菌所生产的那些铁载体,往往没什么别的菌来偷。
这个发现让我有点惊讶——这些病原菌的宿主致病机制千差万别,按理说不该有这么统一的特征?
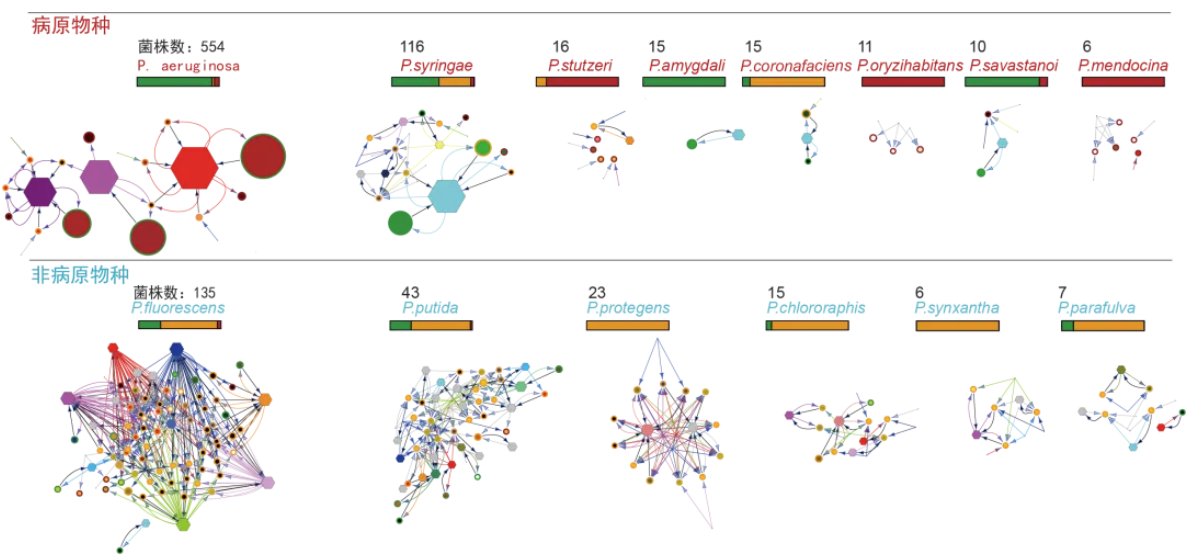
图8. 不同假单胞物种的铁网络对比图。上半部分:病原物种,下半部分:非病原菌物种。每个物种的铁网络上方标注了其菌株数量和物种名,横条中的颜色表示该物种中不同铁行为的分布(绿色:老实;橙色:滑头;红色:蹭饭)。 有趣的是,病原菌(上)的网络结构明显比非病原菌(下)简单,且几乎不见“滑头”。
为了解释这个让人迷惑的现象,我们的起手式就是整个数学模型。这模型也能从动力学上为病原性提供解释,但还是不太直观,每次跟人讨论时都还是要费心地解释:“非生产者”是坏蛋也就罢了,为什么在博弈论上纯粹当贡献者的“单受体生产者”反而更容易成为病原菌?
直到有一次和同行聊天,他提到了"Potluck"这个绝妙的比喻。
这里不得不插播一条解释——这词在中文不好翻译,姑且翻译成“野餐聚会”。毕竟咱们大多数应该生活在一个馋了走两步就能搓一顿的地方。然而,在某些外卖贼贵也不好吃,周围能腿儿到的地方只有subway和吃着并不愉快的pie的地方,饥饿的海外学子们还能怎么办?办个聚会,每个人都带一道菜来,所有人一起分享,这就是potluck。BTW,当年我带的可乐鸡翅惯常惊艳全场——以及有老板会带姜葱龙虾!
接下来,让我们回过头来看看微生物的这场铁的Potluck——就像图 9所显示,用来摄取铁的铁载体就像是微生物所带来的“美食”,而对应的受体像是吃这些食物的“餐具”。对于讲究的食客来说,每种餐具只能用来享用特定的“食物”(筷子吃饺子,叉子吃意面,勺子吃汤圆,也像是我们之前提到的钥匙和锁)。
在这幅插图中,我们可以看到三类不同的微生物参加者:
中间偏左的那群黄色的家伙们看起来其乐融融,这是因为它们都是滑头:每个成员都带来了自己的食物以及对应的餐具,但同时也带了能食用他人食物的“餐具”——你一口我一口,这样才分享得开心嘛。
而匆忙赶来的红色小家伙则是个彻底的“蹭饭者”——它什么食物都没带,却带了一大堆餐具,专门薅别人的吃,谁会喜欢呢?
而右边那个独来独往的绿色“单受体生产者”则采取了另一种方式:它只带了一副餐具,专门用来吃自己带来的独门料理。它既不占别人的便宜,也不让别人占自己的便宜——那你来这party是干什么啊……
在这个比喻里,“老实人”和“蹭饭者”都是这个聚会中的异类——一个拒绝共享,一个拒绝贡献。

图9. 微生物铁元素聚餐会 from @pangshidi
数学模型中,单受体生产者和非生产者的共同特征也都是如此——它们都无法与它人和谐共处。当它们试图入侵一个由别的微生物组成的稳定群落时,要么完全失败,要么就会一举摧毁整个原有群落。反过来,当“小滑头”成为入侵者时,它们一边说着“我不是来拆散这个家,而是来加入这个家的”一边蹑手蹑脚地融入原有群落,不会造成太大的破坏——于是聚会继续。
这个有趣的发现为我们提供了一个潜在的治疗思路:病原菌的“非社交”特性可能是它们的软肋。相比于那些关系网络复杂的“小滑头”们,“老实人”和“蹭饭者”们的铁网络单一得多。这意味着,如果我们能够精准地切断这些特定的铁获取通道,就可能实现对病原菌的靶向抑制,同时又不会过多影响其他有益菌群的铁营养——就像是在Potluck聚会上,只没收某个特定客人的餐具一样。
微生物互作:博弈论的游乐场
当我们谈论病原菌和非病原菌在铁行为上的差异时,实际上触及了一个更深层的问题:微生物世界中“生产”和“盗窃”的博弈。
那么问题来了:微生物也能博弈?它没脑子啊?
那可太能了——反而是我们这样辛苦进化出脑子的灵长目很多时候也潇洒地选择不用——微生物在快速的繁殖和严酷的自然选择中,完美地诠释了进化博弈论:不合适的策略会在短短几天内就被淘汰。
我们认为,铁载体互作系统为我们提供了观察经典博弈论的绝佳窗口。如图 10(1)所示,最简单的是竞争排斥:当两个物种竞争同一种资源时,只会有一个胜利者——这实际导致了一个理论生物学界困惑了80多年的“浮游生物悖论”,但也许这个悖论在天然产物所介导的微生物互作中并不成立——当物种不仅消耗而且产生资源时,经典生态模型的稳定性判据会发生变化[9](铁载体介导的资源分割促进微生物群落的动态共存)——关于这个,我们稍后再聊。
更经典的博弈场景是“公地悲剧”(图 10(2)):当一个纯蹭饭的盗窃者遇到一个老实的纯生产者,前者总能胜出。毕竟,不用付出代价就能获得收益,这种策略怎么看都更有优势。但如果所有菌都这么做,铁载体这种公共资源就会慢慢消失——可它并没有消失。
在经典之外,铁互作网络还给我们展示了一些新颖的博弈场景。比如,当两个“小滑头”相遇时(图 10(4)),它们可能会达成一种“窃取平衡”:你吃我的,我顺你的,搞不好能相安无事。更有趣的是,当更多物种参与这场博弈时,系统的行为会变得更加丰富。在我们最新的预印本研究中,三个“小滑头”之间的互作可以产生类似“剪刀石头布”的周期性震荡(图 10(5)),而五个物种的系统甚至可能进入混沌状态[10]——可这些还只是理论,如果能建立起以铁博弈为模式的微生态实验体系,那好玩的可就多了。
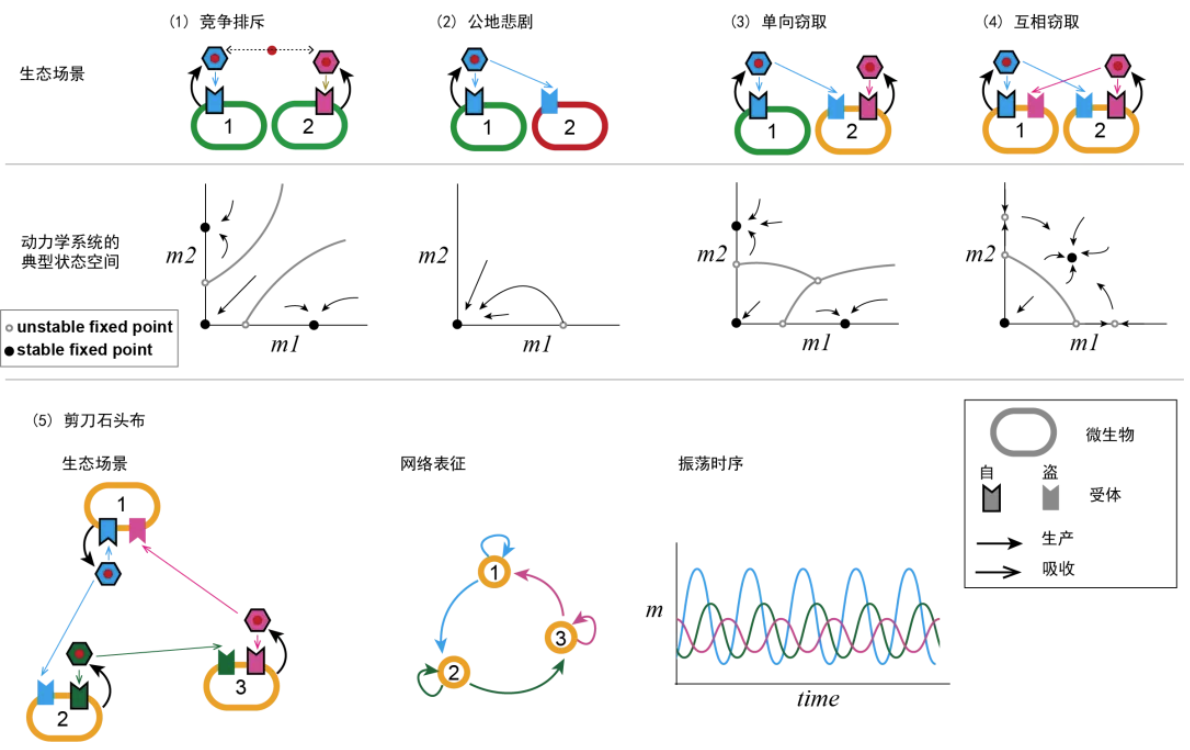
图10. 铁互作中的博弈场景。(1)-(4) 两个物种间的典型互作模式及其动力学行为是(1) 竞争排斥:两个生产者竞争同一资源; (2) 公地悲剧:生产者被盗窃者取代; (3) 单向掠夺:双方都是生产者,但一方能利窃用用另一方的资源;(4) 互相掠夺:两个生产者都互相窃用对方的资源;(5) 更复杂的多物种互作: 左图展示三个物种的互作关系;中图为其网络简化表示;右图显示种群数量随时间的周期性变化,形成类似“剪刀石头布”的动态。
4. “相邻可能性”——永不停歇的化学创新
尽管在生物领域捣腾了这么多年,我还是倔强地觉得自己还在做物理——至少,一些可能有点奇怪的在边边上的物理。
大学时读Stuart Kauffman的书,然后就被《宇宙为家》引到了生物物理里来了。在之后的一本《Reinventing the Sacred: A New View of Science, Reason, and Religion》里,他认为生命系统,特别是它们的进化过程,可能需要一种新的物理学来描述。传统物理学着重于描述已经存在的事物之间的关系和规律,但生命系统不断创造新的可能性,不断扩展自己的状态空间。这个概念特别强调系统在任何时刻都有一些可能的“下一步”,而这些可能性又会开启新的可能性。这种不断前往“相邻可能性”的创新似乎超出了传统物理学的描述框架,却是生命系统的本质特征。
在铁载体的世界里也是如此。每当随机突变出一种新的铁载体结构,它不仅仅是增加了一个新的化学分子,更是开启了一系列新的可能性:新的受体可能进化出来识别它,新的微生物可能开始利用它,新的生态位可能被创造出来——竞争排斥原则的上限因为微生物自己制造化学维度这件事被顶高了。这种化学创新不断推动着可能性的边界,让微生物群落变得越来越丰富多彩。
更有趣的是,这种创新似乎永无止境——NRPS以其极高的代谢代价允许了近乎是无止尽的多样性空间;而演化中的博弈,包括生产者和盗窃者的博弈、和噬菌体与细菌的博弈,推动着系统永远奔向下一个可能——在浩瀚的微生物世界中,还有多少未知的化学创新在等待我们发现?
这也让我们对生命有了新的认识:生命不仅仅是被环境塑造的被动个体,更是不断创造新可能的主动推手。生物通过自己的行为——铁载体、抗生素、光合作用、更复杂的多细胞生物开始修筑堤坝,建造道路和桥梁,飞向天空……不断重塑着它们的生存环境,创造着新的生态位。正是这种永不停歇的创新,让生命的多样性得以绽放。
这个星球上,什么最丰富的?不是阳光;不是氧、硅、铝、和铁;不是微生物;
是可能性。

图11. 微生物次级代谢中永不停歇的化学创新。上图:铁载体-受体配对的演化树。每个新的铁载体(六边形)及其对应的受体(旗形)的出现,都为后续的演化开辟了新的可能性空间,形成分支式的多样化过程。下图:演化驱动下的多样性增长。随着时间推移,新的铁载体-受体配对不断产生,使得整个系统的多样性的理论上限也随之增高。
研究者团队
计算团队:北京大学前沿交叉学科研究院-李志远团队
课题组的研究以“生物系统中的自组织规律”为核心,以数据挖掘和网络动力学等计算方法为手段,着重关注于多细胞体系中模式生成的数学规律,和微生物从演化到群落互作中的定量科学。在微生物生态网络方面,近年来关心的方向为:模块化天然产物的演化规律、结构预测、和人工设计原理;从基因组进化模式推断微生物相互作用网络;微生物互作网络的生态和演化的动力学模型等(https://cqb.pku.edu.cn/zyli/)。

实验团队:南京农业大学LorMe韦中团队
南京农业大学微生态与根际健康实验室依托农业资源与环境国家双一流学科,在国家重点研发计划、973项目、国家自然科学基金(杰青、优青等)和江苏省自然科学基金等项目资助下,在微生态与根际健康、生物肥力、根际免疫、生物污染、噬菌体、农业有机废弃物生物高值化等方面取得系列重要突破。相关工作在Nature Biotechnology、Nature Microbiology、Nature Ecology &Evolution、Nature Communications、Science Advances、PNAS等国际著名期刊发表。

相关文章
1. Gu S, Shao J, He R, Xiong G, Qu Z, Shao Y, Yu L, Zhang D, Wang F, Xu R, Guo P: Forging the iron-net: Towards a quantitative understanding of microbial communities via siderophore-mediated interactions. Quantitative Biology 2025:e84.
2. Li Z, Liu B, Li SH-J, King CG, Gitai Z, Wingreen NS: Modeling microbial metabolic trade-offs in a chemostat. PLoS Computational Biology 2020, 16:e1008156.
3. He R, Gu S, Xu J, Li X, Chen H, Shao Z, Wang F, Shao J, Yin WB, Qian L, et al: SIDERITE: Unveiling hidden siderophore diversity in the chemical space through digital exploration. iMeta 2024, 3:e192.
4. He R, Zhang J, Shao Y, Gu S, Song C, Qian L, Yin W-B, Li Z: Knowledge-guided data mining on the standardized architecture of NRPS: Subtypes, novel motifs, and sequence entanglements. PLOS Computational Biology 2023, 19:e1011100.
5. Gu S, Shao Y, Rehm K, Bigler L, Zhang D, He R, Xu R, Shao J, Jousset A, Friman V-P, et al: From sequence to molecules: Feature sequence-based genome mining uncovers the hidden diversity of bacterial siderophore pathways. eLife 2024, 13::RP96719.
6. Gu S, Shao Z, Qu Z, Zhu S, Shao Y, Zhang D, Allen R, He R, Shao J, Xiong G, et al: Siderophore-receptor coevolution analysis reveals habitat-and pathogen-specific bacterial iron interaction networks. Science Advances 2025.
7. Li Z, Ióca LP, He R, Donia MS: Natural diversifying evolution of nonribosomal peptide synthetases in a defensive symbiont reveals nonmodular functional constraints. PNAS nexus 2024, 3:pgae384.
8. Zan J, Li Z, Tianero MD, Davis J, Hill RT, Donia MS: A microbial factory for defensive kahalalides in a tripartite marine symbiosis. Science 2019, 364:eaaw6732.
9. Shao J, Rong N, Wu Z, Gu S, Liu B, Shen N, Li Z: Siderophore-mediated iron partition promotes dynamical coexistence between cooperators and cheaters. iScience 2023, 26.
10. Shao J, Li Y, Lu J, Gu S, Li Z: Siderophore Piracy Promotes Dynamical Coexistence in Microbial Community. bioRxiv 2023:2023.2011. 2021.568182.