合成生物学为理解生物系统中的复杂基因调控网络以及构建具有特殊功能的工程细胞系统提供了自下而上的方法。尽管合成生物学技术已经可以成功地应用到化工生产、生物医药等领域,但想要实现基因调控回路像电路一样完全可预测设计还有许多问题需要解决。以往已有研究可以实现基因调控回路剂量响应的可预测拼搭,但对于动力学响应还无法实现良好的可预测性。而对于生物系统中某学基因调控回路来说,动力学表达过程决定了其功能,例如适应性回路、振荡回路等等。如何实现基因调控回路良好的动力学可预测性拼搭呢?
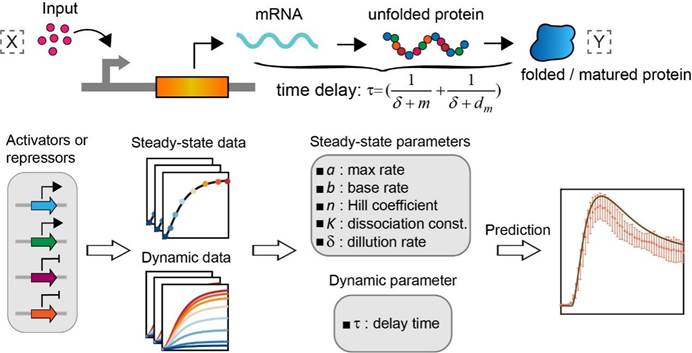
图1 转录、翻译和蛋白折叠等过程会导致一定的时间延迟。利用动力学延迟模型分别拟合剂量响应曲线和动力学响应曲线可以分别得到对应的参数,最后对拼搭得到的基因回路的动力学表达过程进行预测。
以往的研究大多用稳态模型拟合剂量响应曲线得到对应的稳态调控参数,来对后续拼搭的基因调控回路的稳态表达进行定量预测[1]。北京大学定量生物学中心/物理学院的罗春雄课题组通过实验发现仅剂量决定的稳态参数无法对基因回路的动力学表达进行定量描述[2],进而推断是由于转录、翻译和蛋白折叠成熟等中间过程导致了时间延迟(图1),从而使得简单的忽略这些步骤的模型无法准确描述基因调控回路的动力学表达过程。而含转录、翻译和蛋白折叠成熟等步骤的方程组[3]所含的参数过多,拟合实验数据时容易出现过拟合现象。通过细致推导,研究团队得到了一个新的动力学延迟模型(DDM),其稳态决定的部分和延迟相关的部分可以进行分离,而中间细节步骤导致的时间延迟用一个延迟函数概括,并且通过简化可以得到一个特征延迟时间(图1)。该延迟时间仅与mRNA降解速率、蛋白折叠成熟速率和细胞生长速率相关。研究团队通过一步调控实验,换不同成熟时间的荧光报告蛋白[4]作为指示,发现其剂量响应可以用一组参数描述,而延迟时间有明显的不同,证明了动力学延迟模型的可靠性。
之后研究团队提出了通过两步调控回路反推中间调控元件的参数的方法,用以获得任意非标记的调控蛋白的所有参数,这些参数包括剂量决定的稳态参数和延迟时间参数等。实验中通过构建一系列含不同激活子和抑制子的两步调控回路,用已经标定好的荧光蛋白参数结合动力学延迟模型,通过分别拟合剂量响应曲线以及动力学响应曲线得到了8个激活子和5个抑制子的调控参数(图1)。
最后,研究团队利用参数刻画完成的调控元件拼搭了三种不同的基因调控回路,包括简单的三步调控回路和两个结构不同的不一致前馈回路,发现动力学延迟模型相对于传统不考虑延迟的模型可以更加准确地描述基因调控回路的动力学表达过程(图2)。
工作中开发的实验与理论方法可以广泛的应用于获得元件的动力学参数以及进行各类具有动力学特性的基因回路的预测中。

图2 动力学延迟模型模拟(DDM)、不含延迟模型模拟(Protein-only model)与实验对比。
总结:工作理论推导得到了动力学延迟模型,并给出了由于转录、翻译和蛋白折叠成熟等过程导致的时间延迟的公式。并且提出了通过两步调控回路实验结合模型获取中间调控元件参数的方法,可以通过时序数据与理论模型得到各类调控元件的参数;最后工作还构建三个不同的基因调控回路,验证了无自由参数的合成基因调控回路的动力学可预测性拼搭。该工作于2024年3月15日发表于学术期刊《iScience》,题目为“The dynamic-process characterization and prediction of synthetic gene circuits by dynamic delay model”。链接: https://doi.org/10.1016/j.isci.2024.109142。
文章第一作者为北京大学物理学院博士研究生孙燕红,通讯作者为北京大学定量生物学中心/物理学院罗春雄教授及国科温州研究院博士后张凤宇,参与人包括欧阳颀院士。该工作得到国家重点研发计划项目、国家自然科学基金项目、北京大学定量生物学中心、物理学院人工微结构和介观物理国家重点实验室、国科温州研究院的支持。
1. Y. Zong, H.M. Zhang, C. Lyu, et al., Insulated transcriptional elements enable precise design of genetic circuits. Nat Commun, 8 (1), 52 (2017).
2. Y. Sun, F. Zhang, L. Li, et al., Two-Layered Microfluidic Devices for High-Throughput Dynamic Analysis of Synthetic Gene Circuits in E. coli. ACS Synthetic Biology, 11 (12), 3954-3965 (2022).
3. M. Tomazou, M. Barahona, K.M. Polizzi, et al., Computational Re-design of Synthetic Genetic Oscillators for Independent Amplitude and Frequency Modulation. Cell Syst, 6 (4), 508-520 e5 (2018).
4. E. Balleza, J.M. Kim, and P. Cluzel, Systematic characterization of maturation time of fluorescent proteins in living cells. Nat Methods, 15 (1), 47-51 (2018).