撰文 | FCB
面对环境压力,细胞需要做出正确的应激响应[1]。在应激响应过程中,转录因子常占据核心位置[2]。近期在单细胞水平上的研究发现,应激响应过程中,转录因子常常展示出复杂的动力学行为,比如脉冲式的进出核动力学,这些动力学行为常具有随机性和重复性[3]。不仅如此,不少应激响应过程同时受到数个具有动力学特征的转录因子的组合调控,这些转录因子的动力学关系如何调控基因表达并影响细胞命运成了理解应激响应的关键。
芽殖酵母Saccharomyces cerevisiae中,许多压力响应相关的转录因子具有脉冲动力学行为[4],其中,最具代表性的是Msn2和Msn4。Msn2和Msn4负责调控对于多种环境压力的一般应激响应(general stress response),并共同调控超过200个目标基因[5]。然而,这两个转录因子的动力学关系如何影响细胞的压力生存能力仍有待研究。不仅如此,作为一对同源转录因子,Msn2和Msn4在进化中得到了保留,其动力学特征在此过程中的潜在功能也不得而知。
近日,北京大学前沿交叉学科研究院定量生物学中心林一瀚课题组、北京大学数学科学学院邓明华课题组合作在Nature Communications发表了题为“Yeast cell fate control by temporal redundancy modulation of transcription factor paralogs”的文章。通过单细胞显微成像、转录组分析、数学建模等方法,研究人员揭示了酵母细胞中基于Msn2和Msn4动力学的命运调控机制,即 “时序冗余性调控”,并探讨了该机制在进化上的潜在功能。

首先,研究人员发现Msn2/Msn4动力学的脉冲时序关系受到环境压力的调控。随着环境压力强度的增大,Msn2/Msn4脉冲的重合率降低,即这两个转录因子倾向于分开单独脉冲。利用实时转录报告系统及转录组分析,发现Msn2/Msn4在单个脉冲层面上具有功能冗余性。据此,推测当Msn2/Msn4脉冲重合率降低时,分开的脉冲对于下游基因的激活能力更强,进而提升细胞在压力下的生存能力。进一步的RNA-seq及单细胞显微成像实验结果支持了这一推断。以上数据揭示,细胞通过改变功能冗余的Msn2/Msn4脉冲来动态调控它们在下游基因激活上的冗余性,进而动态调控基因表达,并增强细胞在压力下的生存能力。研究人员将这一机制称为“时序冗余性调控”(图1),进一步利用数学建模,提出了基于翻译后修饰的“时序冗余性”调控机制,并通过实验初步验证了该机制。
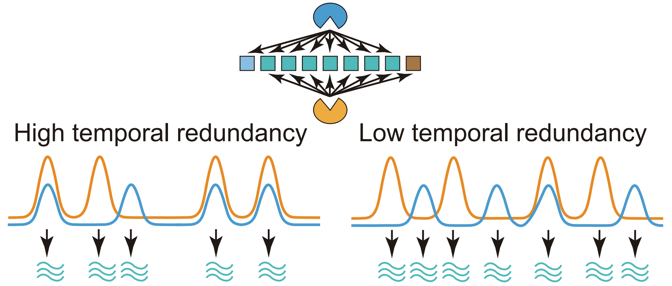
图 1 “时序冗余性调控”可以增大目标基因激活的动态范围
研究人员进一步提出了基于“时序冗余性调控”的同源转录因子功能冗余性保留假说,即转录因子动力学可能有助于维持在进化过程中Msn2/Msn4的冗余功能。通过实验分析其它酵母,发现在Candida glabrata中,Msn2/Msn4同源蛋白间也具有相似的功能冗余性,且两转录因子的动力学关系也受到调控,暗示着“时序冗余性调控”在进化中的潜在功能(图2)。此外,研究人员利用公共单细胞数据分析了人源转录因子的动力学特征,并探讨了上述机制潜在的广泛意义。
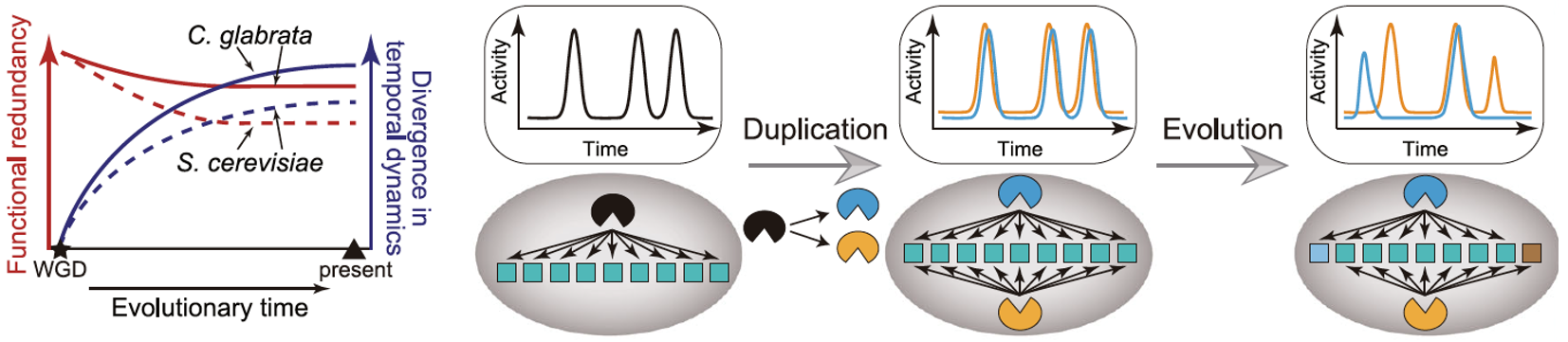
图 2 “时序冗余性调控”在进化上的潜在功能
综上,该研究发现了芽殖酵母细胞中基于同源转录因子动力学时序关系调控的应激响应及命运控制机制,并探讨了这一机制在同源转录因子功能冗余性进化上的潜在意义。
北京大学前沿交叉学科研究院定量生物学中心林一瀚研究员为本论文通讯作者,前沿交叉学科研究院博士研究生吴岩为第一作者,北京大学数学科学学院邓明华教授为该研究提供了关键指导,前沿交叉学科研究院博士研究生吴佳奇参与了该研究工作。该研究得到了国家重点研发计划、国家自然科学基金以及北大-清华生命科学联合中心的资助。北京大学定量生物学中心成像平台和微流平台,以及北大-清华生命科学联合中心高性能计算平台为该研究提供了实验与计算资源。
原文链接:https://www.nature.com/articles/s41467-021-23425-0
参考文献
1. Kultz, D., Molecular and evolutionary basis of the cellular stress response. Annual Review of Physiology, 2005. 67: p. 225-257.
2. Purvis, Jeremy E. and G. Lahav, Encoding and Decoding Cellular Information through Signaling Dynamics. Cell, 2013. 152(5): p. 945-956.
3. Levine, J.H., Y. Lin, and M.B. Elowitz, Functional roles of pulsing in genetic circuits. Science, 2013. 342(6163): p. 1193-200.
4. Dalal, C.K., et al., Pulsatile Dynamics in the Yeast Proteome. Current Biology, 2014. 24(18): p. 2189-2194.
5. Estruch, F., Stress-controlled transcription factors, stress-induced genes and stress tolerance in budding yeast. FEMS Microbiol Rev, 2000. 24(4): p. 469-86.