细胞生活在不断变化的复杂环境中,通过信号转导通路传递外部的信号到细胞内的调控网络。2023年10月26日,北京大学物理学院/定量生物学中心李方廷研究组在PNAS Nexus在线发表了名为“Multiple activation of the yeast S-phase checkpoint are Poisson processes”的文章[1],结合芽殖酵母的不同突变株的单细胞实验结果和数学模型,发现了S期检查点的多次激活过程是泊松过程,揭示了酵母细胞在噪声作用下可能产生的复杂动力学行为。
在DNA复制压信号的作用下,S期检查点信号转导通路激活,修复DNA损伤并恢复DNA的复制进程[2]。当芽殖酵母细胞受到化学药物羟基尿(HU)介导的DNA复制压力刺激时,S期检查点的关键激酶Rad53将被磷酸化,从而激活S期检查点。我们前期的研究发现,在相同的DNA复制压力下,酵母中S期检查点的激活表现出细胞间的异质性;其中在一个细胞周期过程中,由Rad53磷酸化引起的S期检查点正反馈环路构成一个双势阱,生化反应促进了S期检查点的首次激活过程为跨越势垒的越障过程[3]。然而,S期检查点通路的恢复过程还包含一个负反馈环,所以在一个细胞周期过程中,S期检查点可能会被多次激活[4]。一般来说,同时具有正反馈回路和负反馈回路的网络可能会表现出振荡行为,例如p53和NF-kB信号转导途径[5]。然而,在生物噪声下,芽殖酵母S期检查点可能会表现出更复杂的动力学特性。
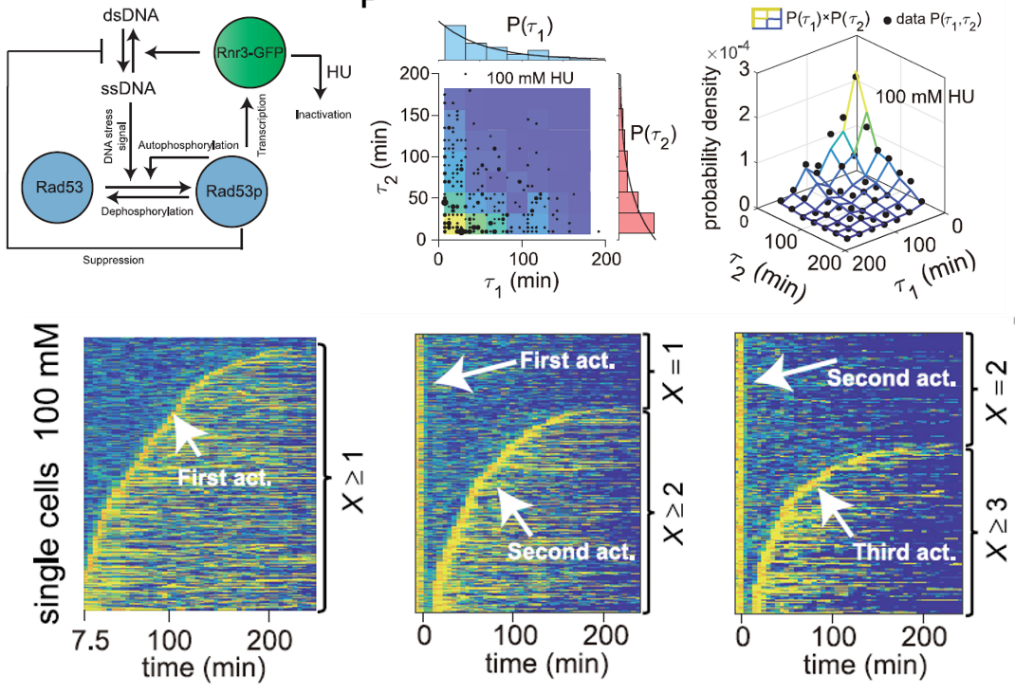
图1 左图:芽殖酵母S期检查点通路示意图。右图:S期检查点多次激活的等待时间独立同分布。下图:S期检查点通路的泊松过程激活热图。
通过单细胞实验和分析,我们发现了酵母检查点通路中的多重激活过程为泊松过程。我们在S期检查点核心激酶Rad53下游蛋白Rnr3后融入绿色荧光蛋白GFP,并且通过GFP的变化率来表征S期检查点的激活情况。通过统计GFP变化率的分布,我们发现S 期检查点的细胞间变异不仅在于激活时刻,还在于单个细胞周期内的激活事件次数。通过计算多次激活的速率以及联合分布特征,我们证明了S期检查点每次激活的等待时间是独立且同分布的指数分布,即多次激活构成了一个泊松过程(图1)。我们称之为泊松激活过程(Poisson activation process),这一现象再次证实了酵母S期检查点激活存在显著的随机性。
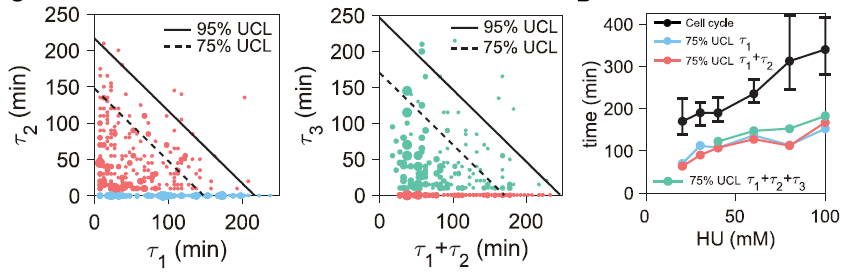
图2 不同激活次数细胞的等待时间上限。
进一步,我们发现了S期检查点的泊松过程激活存在一个稳定的时间上限。对于不同激活次数的细胞群而言,其等待时间均存在一个上限。这个时间上限在不同激活次数的细胞群中均相同,仅与HU浓度正相关。考虑到S期检查点激活时,DNA会停止解旋,我们认为泊松激活过程等待时间的上限就代表了DNA的复制时间。DNA的复制时间构成了泊松激活过程中的时间上限,S期检查点的激活事件是在DNA复制时间内的泊松过程。
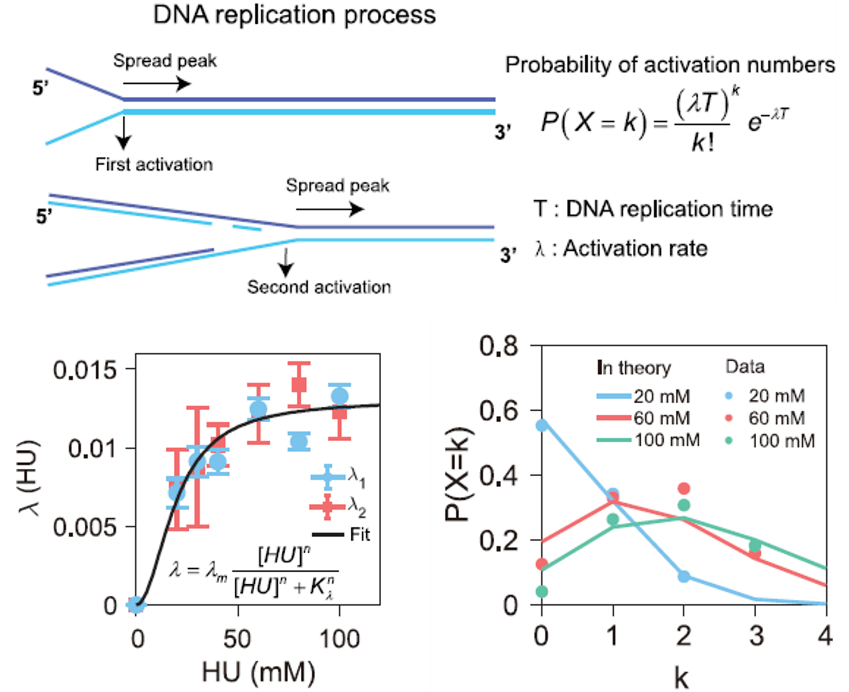
图3 S期检查点泊松激活过程模型。上图:S期检查点多次激活示意图。左图:激活速率与HU浓度的关系。右图:一个细胞周期内激活次数的泊松分布。
在酵母细胞中,单个细胞周期中S期检查点的激活事件的数量也遵循泊松分布。以 HU 为参数的简单泊松过程模型可以很好地描述野生型(WT)和突变株的激活事件数量,模型中HU浓度作为参数同时影响了激活速率和DNA的复制速度。如图3所示,一个细胞周期内S期检查点的激活次数很好的服从了泊松分布。总体而言,所观察到的泊松激活过程的动态变化与其他经过深入研究的信号转导途径不同。
这项研究通过数学物理模型分析了酵母单细胞实验,揭示了生物过程的动态机制。作者为北京大学高鑫、周沛劼和李方廷。该研究得到了国家自然科学基金等机构的支持。
参考文献
[1] Gao, X., Zhou, P., & Li, F. (2023). The multiple activations in budding yeast s-phase checkpoint are poisson processes. PNAS Nexus, pgad342.
[2] Segurado, M., & Tercero, J. A. (2009). The S-phase checkpoint: targeting the replication fork. Biology of the cell, 101(11), 617–627.
[3] Zhou, P., Gao, X., Li, X., Li, L., Niu, C., Ouyang, Q., ... & Li, F. (2021). Stochasticity triggers activation of the s-phase checkpoint pathway in budding yeast. Physical Review X, 11(1), 011004.
[4] Clémenson, C., & Marsolier-Kergoat, M. C. (2009). DNA damage checkpoint inactivation: adaptation and recovery. DNA repair, 8(9), 1101-1109.
[5] Hafner, A., Bulyk, M. L., Jambhekar, A., & Lahav, G. (2019). The multiple mechanisms that regulate p53 activity and cell fate. Nature reviews Molecular cell biology, 20(4), 199-210.