自2012年George Church团队首次在DNA分子中实现较大规模的数据存储以来,下一代DNA数据存储已经走过了十年的历程。在此期间,混合了大量DNA短链的DNA池一直作为主流的存储载体,与之相配套的信息写入方法是阵列DNA合成,信息读出方法是高通量短读长测序(即下一代DNA测序技术,例如Illumina)。这种实施方式有效地实现了大规模数据存储,以及对于DNA池中特定小文件的随机访问。然而,这种存储方式通常只适用于存储时间长但读取频次较低的数据(称为“冷数据”),而对于规模不大但读取和分发需求较高的数据则不十分适用。为了扩展DNA数据存储的用户群体、促进完整的DNA数据存储生态系统的建立,需要在使用DNA池进行集中式大规模数据存储的同时,开发便携式的数据存储系统。
2023年2月3日,北京大学定量生物中心钱珑/欧阳颀团队在Advanced Science在线发表了题为“Mobile and Self-sustained Data Storage in an Extremophile Genomic DNA”的研究论文,该研究在细菌中建立了一套便携式、可自我维持的DNA数据存储系统,用来实现双模式存储、动态数据维持、快速信息检索和稳健的数据恢复。

在该研究中,作者首先开发了一套通用型的基因组整合工具箱(RSGE toolbox),从而以genome landing pad的方式,将较大片段的DNA整合到不同细菌之中。为了验证这一工具箱的有效性,编码了人工信息的DNA片段(长度为数十kb)被分别整合到模式生物E. coli和一种嗜盐菌Halomonas bluephagenesis的基因组中。虽然这两个物种在分子进化树上亲缘关系较远,但利用前述工具箱均实现了有效的DNA整合,表明了其泛用性。
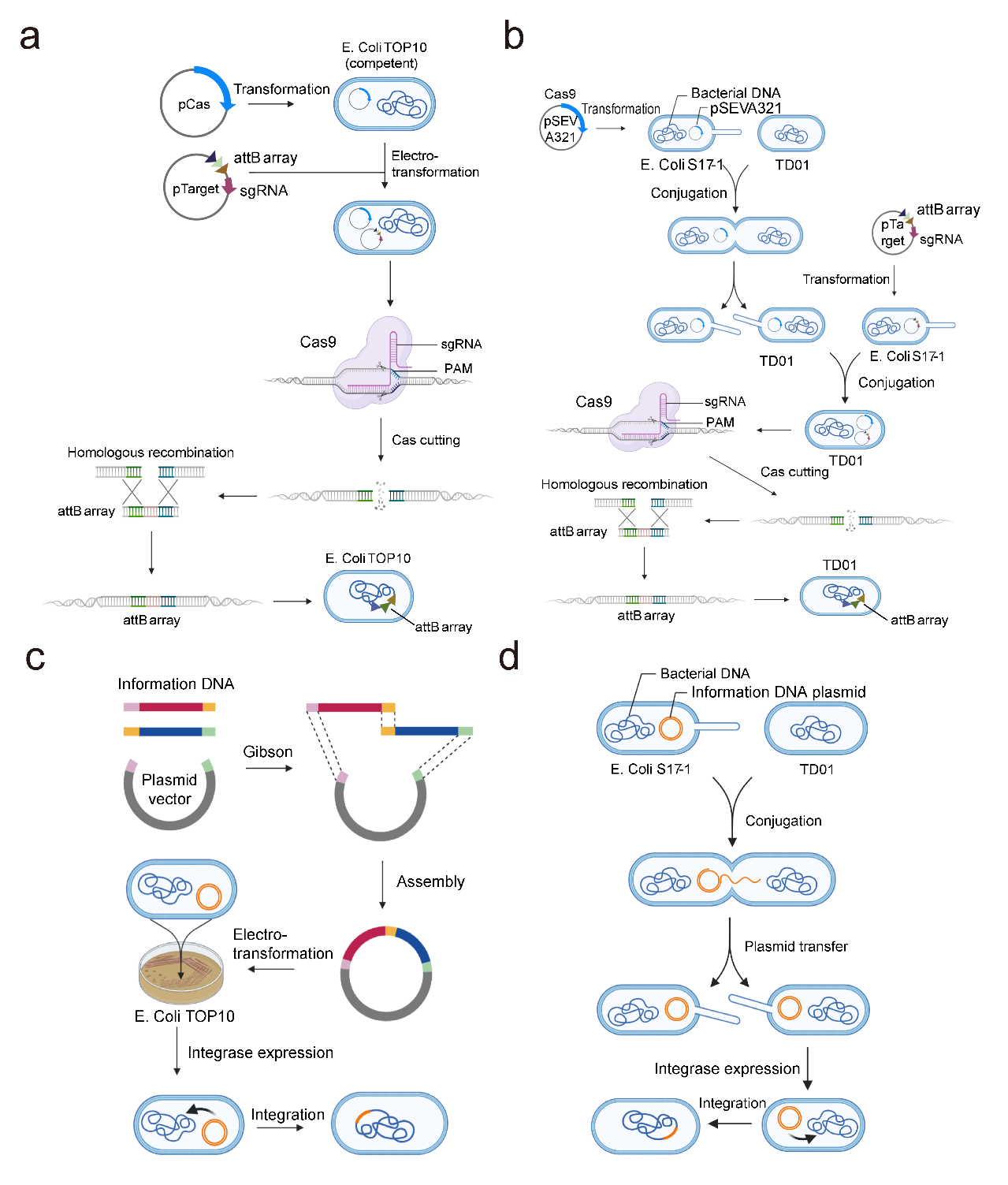
图1 使用RSGE工具箱,将存储人工信息的DNA片段整合到细菌基因组中。
之后,作者对基因组中整合了信息DNA的菌株进行了传代实验,并在此期间不断监测信息DNA的完整性。在100天的时间内,细菌分裂至第2000代,未发现信息DNA丢失或重组,仅有少量点突变。该菌株被置入家用冰箱(-20℃)中保存超过一年后,仍可在适宜的条件下重新恢复生长,并且其中的信息DNA未发生丢失。以上结果表明,使用RSGE工具箱整合了信息DNA的菌株可以在活跃生长/长期休眠两种模式下维持其基因组中信息DNA的完整性。
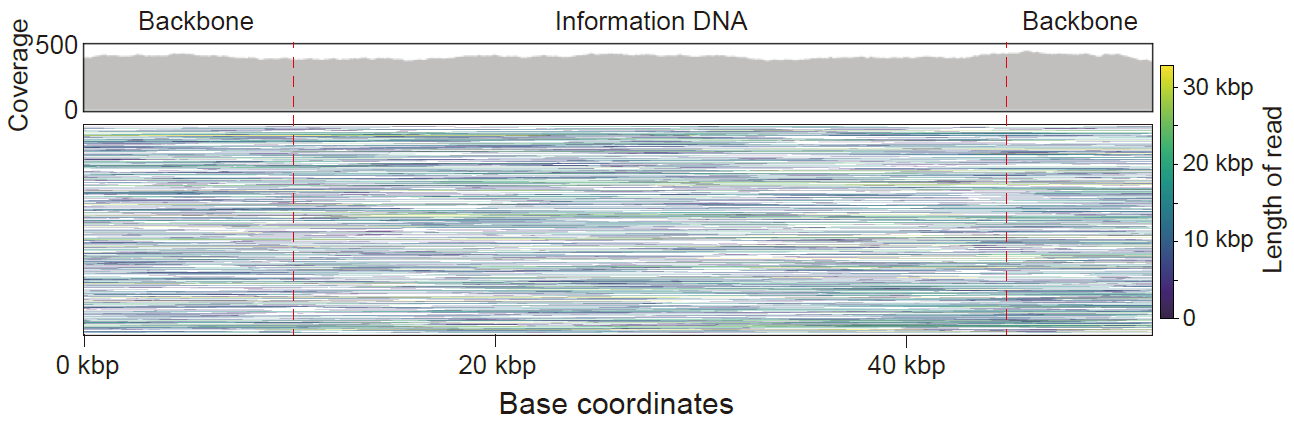
图2 纳米孔测序中的单read分析与总体覆盖率。
作为一种极端微生物,H. bluephagenesis由于其嗜盐特性,具有对生物污染的天然抗性,这对于构建便携式数据存储系统是一个优势。为了验证这一点,作者将整合了信息DNA的H. bluephagenesis和E. coli分别在无菌和开放的环境中培养18小时,之后检索其中的信息,发现H. bluephagenesis在两种状态下均可以实现信息恢复,而E. coli仅在无菌培养条件下可以实现信息恢复。
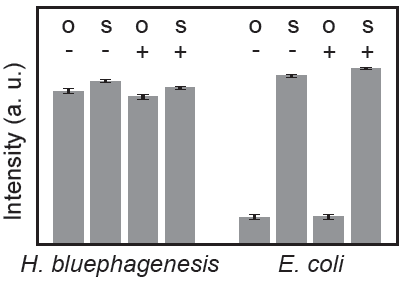
图3 从无菌/开放培养的H. bluephagenesis和E. coli中恢复信息。
基于以上结果,作者使用H. bluephagenesis搭建了一个桌面式数据存储系统。整合了信息DNA的菌株被转移到液体培养基中,然后放置在开放的容器中自发生长,不施加温控、震荡等专业培养条件。每隔一定时间,取出一定体积的培养基用于信息恢复,同时补充同等体积的纯净培养基。结果表明,在取样间隙,细菌的生长和分裂自发地补足了取样的损耗,保证了信息多次分发和读取而不耗竭。这一系统不依赖于任何专业人员或复杂设备,可以在普通家庭环境或野外环境中使用。
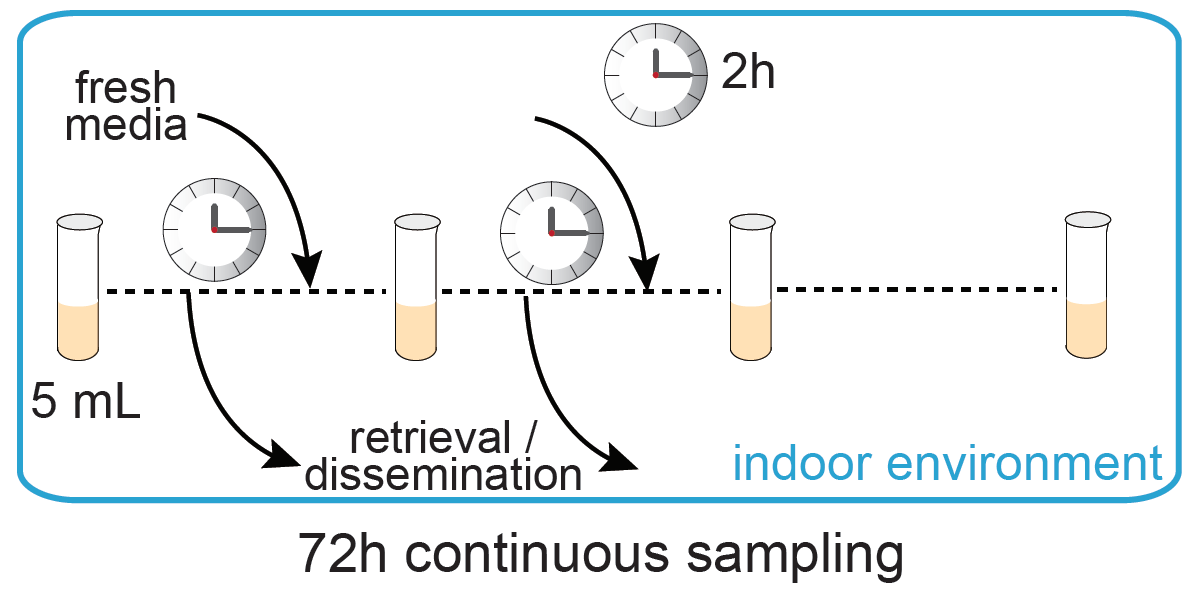
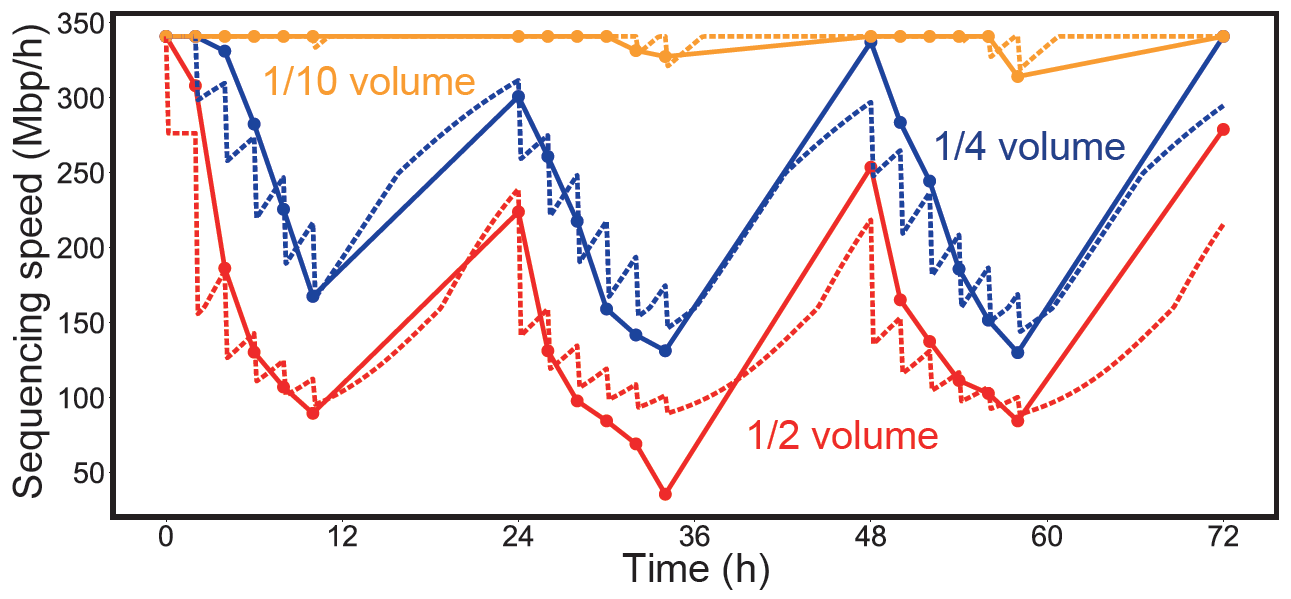
图4 使用桌面式数据存储平台进行频繁的信息分发和检索。
为了实现快速、便捷的信息读取,需要使用便携式纳米孔测序仪,但其测序错误率较高,可达10-15%,这极大地影响了信息的可靠性。为了应对这一问题,作者开发了一种新型的DNA纠错编解码算法(MEPCAL),通过使用了嵌套的RS码和RaptorQ码,并引入了序列锚定策略,从而有效地应对纳米孔测序中各种类型的错误。使用MEPCAL编码的信息DNA的纳米孔测序结果表明,仅仅使用覆盖率小于10×的数据量就可以无损地恢复原始信息。
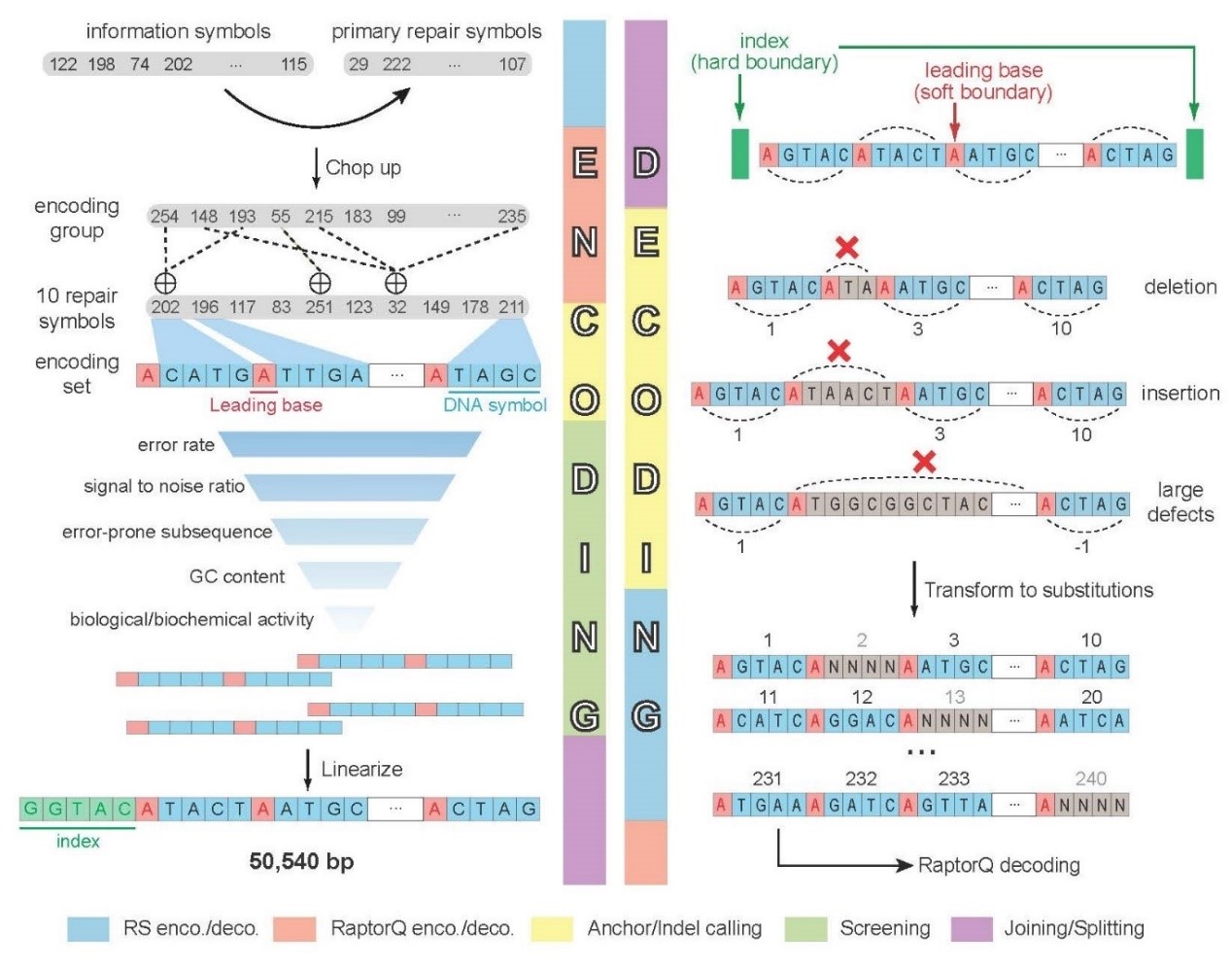
图5 任意长度混合错误处理编码(Mixed Error Processing Coding for Arbitrary Length)。
最后,作者基于MEPCAL开发了一个DNA编码的数学模型,以指导在不同的预期序列错误率下选择参数组合,来实现编码密度和解码稳定性之间的最优权衡。使用这一模型的预测结果,对更大规模的数据进行了编码测试,结果表明MEPCAL可以被应用于大规模数据存储而不会出现显著的性能衰退。上述结果表明,MEPCAL具有优良的稳健性和可扩展性,可以满足实际应用的需求。
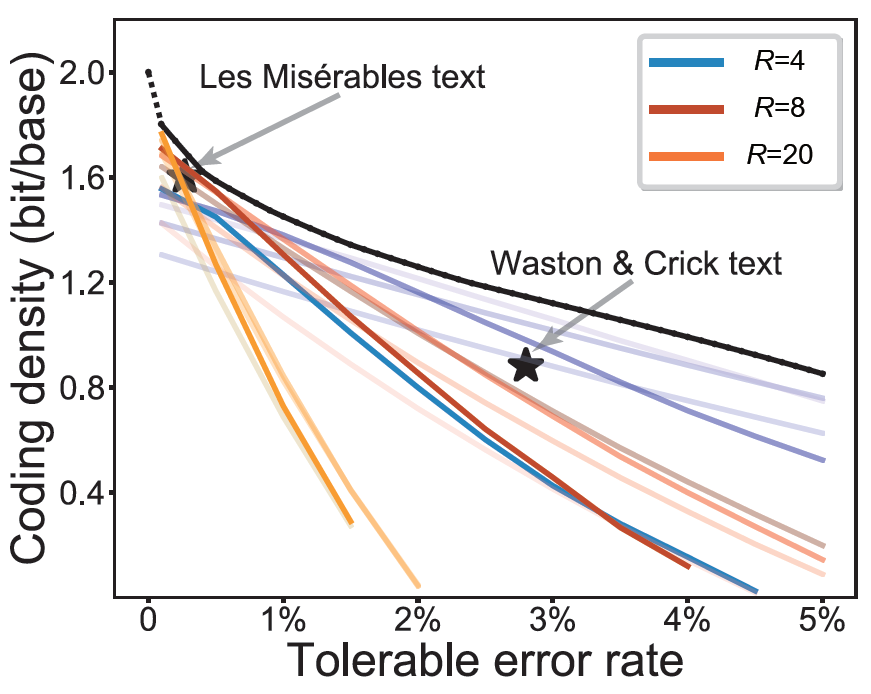
图6 DNA编码的定量模型与本研究中获得的编码密度-稳定性权衡。
总结而言,该研究开发了一种便携式DNA数据存储系统,以补足DNA池在特定应用场景中的不足。这种便携式系统是用户主导的,用户可以在一定程度上对存储在DNA中的数据进行操作,例如信息的复制、分发和读取,而不需要专业人士的介入和复杂设备的支持。将集中式存储和分布式存储联合起来,将有利于实现对各种数据的全方位存储,建立完整的DNA数据存储生态系统。
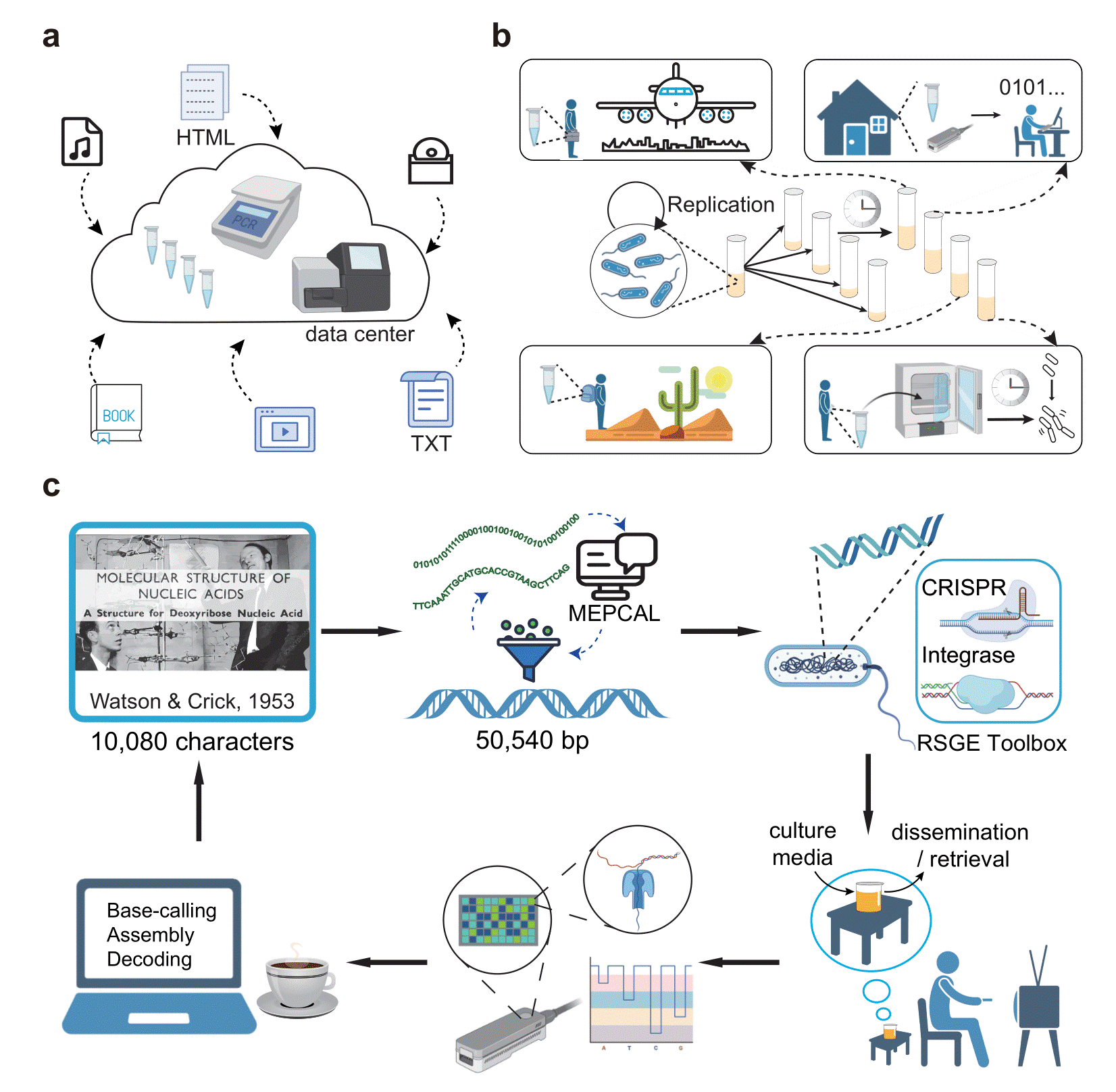
图7 研究总结
北京大学定量生物学中心博士研究生孙法家和博士后董一名(现为芯宿科技CSO与联合创始人)为该论文共同第一作者,北京大学定量生物学中心钱珑和欧阳颀教授为该论文的通讯作者。华大智造高级副总裁、青岛华大智造总经理倪鸣博士和深圳华大生命科学研究院平质副研究员、孙宇辉为该论文的共同作者。本研究得到科技部重点研发计划和国家自然科学基金的支持。
相关阅读
NSR综述:DNA信息存储的现状与未来
https://mp.weixin.qq.com/s/zYjoSI_Oe4cvmIzzsOnIgw
DNA数字信息存储:造梦、追梦与圆梦
https://mp.weixin.qq.com/s/3OsD6iknj4HE54VMqAPFUQ
【特约评述:钱珑】DNA数字信息存储的研究进展
https://mp.weixin.qq.com/s/hGk2vQt8MHzv9edzpQifiw