蛋白质作为生命活动的主要执行者,其功能的发挥往往伴随着复杂的构象转变。理解这些转变的机制,以及蛋白质(尤其是膜蛋白)如何与周围环境(如脂质)相互作用,对揭示生命奥秘、理性设计新型药物至关重要。全原子分子动力学(All-atom MD)模拟虽然能提供原子级别的动态细节,但其高昂的计算成本和有限的模拟时间尺度,使其往往难以捕捉蛋白质在生理条件下的全局构象变化(通常在毫秒量级以上)。粗粒化(CG)Gō-like模型虽然能加速模拟过程,但传统的隐式溶剂模型无法准确描述蛋白质与脂质等显式环境的相互作用。
为应对上述挑战,北京大学定量生物学中心宋晨团队致力于开发高效且能兼顾环境效应的蛋白质动力学模拟方法。该团队前期在《Journal of Chemical Theory and Computation》(JCTC)上发表的 Switching Gō-Martini 方法(JCTC 2024, 20, 2618-2629),将经典的“切换Gō模型”思想与Martini 3粗粒化力场相结合,通过在已知的两个构象态之间切换势能面,成功模拟了多种蛋白质(包括GlnBP、AdK、β2AR以及膜蛋白P4-ATPase家族脂质翻转酶ATP8A1-CDC50a)的大规模构象转变,并首次揭示了ATP8A1-CDC50a转运底物脂质分子的跨膜动态过程。Switching Gō-Martini 方法在保持计算效率的同时,实现了在显式环境下对蛋白质构象转变及其对脂质互作影响的探索。
然而,Switching Gō-Martini方法仍存在一些局限,如需要手动切换势能面,导致转变过程非自发、可能引入非合理路径等。为进一步突破这些限制,宋晨团队近期在JCTC上发表了题为“Multiple-Basin Gō-Martini for Investigating Conformational Transitions and Environmental Interactions of Proteins”的研究论文,报道了新一代的 Multiple-Basin Gō-Martini方法。 该模型通过引入指数形式的耦合方法,构建了包含多个势阱的更加真实的蛋白质势能面,使得蛋白质能够在不同构象态之间自发转变(图1)。 Multiple-Basin Gō-Martini方法不仅显著提升了模拟的真实性,还能提供关于转变动力学、热力学和中间态的半定量信息,为深入理解蛋白质构象转变机制及其与脂质等环境的相互作用开辟了新途径。
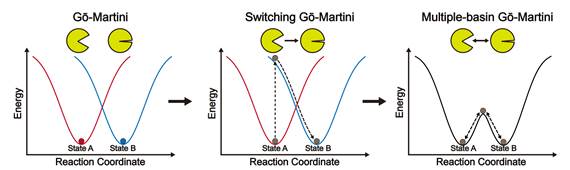
图1:Gō-Martini模型发展示意图。
为验证Multiple-Basin Gō-Martini方法的准确性并评估其性能,研究团队选取了五种具有代表性的蛋白质体系(GlnBP, Arc, Hinge, SemiSWEET以及TRAAK)进行测试。这些体系涵盖了可溶性蛋白与膜蛋白,结构域集体运动与二级结构转变,天然蛋白与从头设计蛋白,以及普通体系与膜表面张力体系等。研究结果表明,Multiple-Basin Gō-Martini不仅能准确再现各类蛋白质的构象转变路径并捕获关键中间态,而且相较于隐溶剂Gō-like模型,还能深入揭示环境因素(特别是蛋白质构象转变过程中的蛋白-脂质相互作用)对蛋白质状态分布的调控机制(图2)。特别地,该方法首次完整模拟了机械敏感离子通道TRAAK在近生理表面张力(~10 mN/m)条件下的"Down-Up"状态转变过程。研究发现当TRAAK处于向下状态时,脂质分子的极性头部(而非疏水尾部)选择性地插入由TM2和TM4螺旋形成的窗口中;当蛋白质从向下状态转为向上状态时,脂质分子呈现从窗口逐渐离去的动态过程。这些结果展示了Multiple-Basin Gō-Martini方法在模拟蛋白质构象转变,尤其是膜蛋白动态转变过程中的蛋白-脂质相互作用的独特优势。
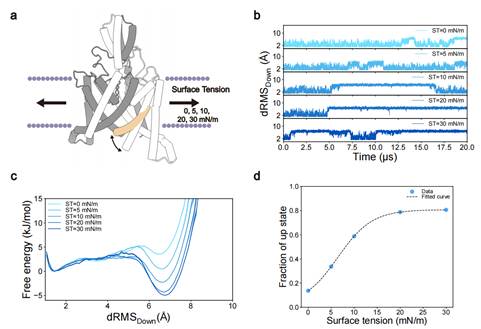
图2:不同表面张力对TRAAK构象分布的影响。
目前,研究团队正致力于将Multiple-Basin Gō-Martini方法与更高分辨率的模拟技术(All-atom MD)和增强采样方法相结合,以期获得更定量的蛋白质能量景观信息。同时,利用该方法研究多蛋白的协同工作机制,以及为基于机器学习的蛋白质构象预测模型提供高质量的转变路径训练数据,也是未来的重要发展方向。宋晨团队发展的Gō-Martini系列方法为研究复杂生物系统中的蛋白质动态行为提供了一套新的有效工具。团队已将该方法相关的代码和教程在GitHub上开源(链接:https://github.com/ComputBiophys/CTGoMartini),方便相关研究领域社区的使用和进一步开发。
北京大学-清华大学生命科学联合中心博士毕业生、现福州大学医学院副教授杨嵩为该论文的第一作者。北京大学定量生物学中心及北京大学-清华大学生命科学联合中心宋晨研究员为论文的通讯作者。本研究得到了国家重点研发计划和国家自然科学基金创新研究群体项目的资助。分子动力学模拟工作在北京大学生命科学联合中心高性能计算平台“北极星”完成。
原文链接:https://doi.org/10.1021/acs.jctc.5c00256