细胞极化广泛存在于原核与真核生物,指特定分子在细胞质内或细胞膜上形成空间不对称分布 [Development, 2011];这一过程使细胞获得空间取向,对其分裂、分化、迁移、以及伤口愈合等微观-宏观现象至关重要 [Science, 2000; Nature, 2002]。例如,在胚胎发育过程,极化的母细胞可以将空间不对称分布的分子及其下游分子不均匀地分配到两个子细胞内,使其获得不同的体积和命运 [Nat. Rev. Mol. Cell Biol., 2004];在多细胞层面,细胞的集体极化可以调控器官有序发育和抑制肿瘤 [Science, 2000]。然而,细胞如何在复杂多变的生物环境中实现分子稳定的空间不对称分布,维持细胞极性,是一个基础的生物学、物理学问题。
近日,北京大学汤超实验室、香港浸会大学/松山湖材料实验室汤雷翰实验室在eLife在线发表文章Balancing reaction-diffusion network for cell polarization pattern with stability and asymmetry [https://doi.org/10.7554/eLife.96421.3],报道了反应扩散网络结构和参数的整体平衡是细胞极性斑图稳定的关键因素。早在1988年,Kemphues等人发现PAR蛋白家族介导了秀丽隐杆线虫受精卵的极化过程 [Cell, 1988],随后一系列的实验证明位于前部的aPARs (anterior PAR proteins) 与位于后部的pPARs (posterior PAR proteins) 相互拮抗网络构成了细胞极化的分子基础 [Development, 2003]。2008年,Tostevin等人对该二节点拮抗网络构建物理模型,理论证明了该反应扩散网络对线虫受精卵极化的生物物理原理 —— 即两组拮抗蛋白能够在细胞质(快扩散)和细胞膜(慢扩散)分别存在,且结合细胞膜后能抑制另一蛋白的结合,最终形成空间分离(图1 - 第1列) [Biophys. J., 2008];后来,2012年,汤超实验室与合作者针对细胞极化穷举了所有二节点以内的网络结构,鉴定出包括二节点拮抗网络在内的有效网络结构,并基于计算预测在酿酒酵母中人工合成了新的极化细胞 [Cell, 2012]。近年来,其他参与极化过程的蛋白(CDC-42、LGL-1、CHIN-1等) 逐渐被发现 [Mol. Biol. Cell, 2010; Development, 2010; Dev. Cell, 2015],细胞极化如何在更多节点的现实网络中存在,以及具体功能属性(稳定性、界面位置、生物物理参数鲁棒性等)如何被设计,是一个近年备受关注的实验与理论问题 [Cells, 2020; Cell Rep., 2021]。本工作从传统的二节点网络出发,关注网络结构和参数如何影响细胞极化稳定性和界面,并将其应用到线虫的五节点网络中探索它隐藏的进化优势,探究普适的细胞极性网络设计原理。
1. 细胞极化的反应扩散模型
研究团队首先基于反应扩散方程在一维空间内搭建了模拟细胞极性的物理模型,该模型能适配由各种节点和调控关系组成的网络(图1)。分子X在细胞膜上的空间浓度 可以被数学描述为:
可以被数学描述为:
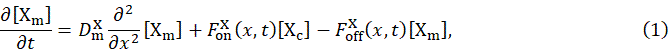
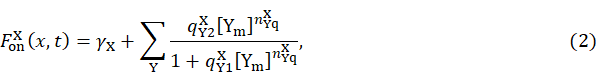
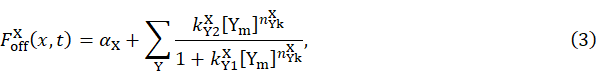
其中, 表示分子X在细胞膜上的扩散系数;
表示分子X在细胞膜上的扩散系数; 包含分子X的基础结合率
包含分子X的基础结合率  ,以及其他蛋白(或者自己)对它的促进作用(用参数为
,以及其他蛋白(或者自己)对它的促进作用(用参数为  的希尔方程表示);
的希尔方程表示); 包含分子X的基础解离率
包含分子X的基础解离率  ,以及其他蛋白(或者自己)对它的抑制作用(用
,以及其他蛋白(或者自己)对它的抑制作用(用  的希尔方程表示) [Cells, 2020]。
的希尔方程表示) [Cells, 2020]。
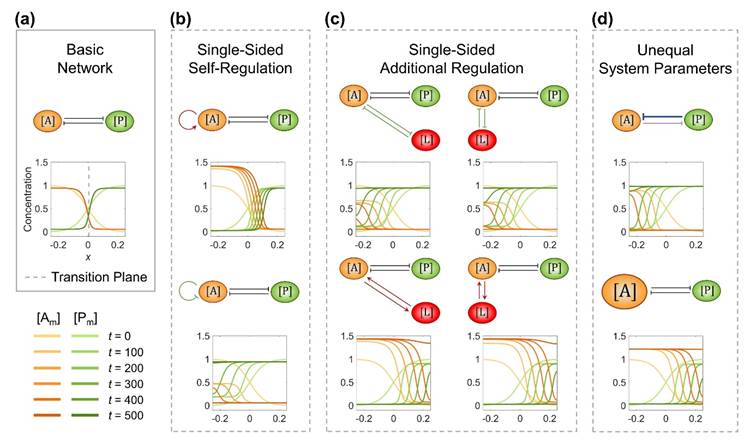
图1. 在对称的二节点网络中引入单一调控(单边自我调控、单边额外调控、不对称系统参数)时,斑图界面将持续往一侧移动,最终导致细胞极性崩塌。
2. 简单二节点网络的平衡原理和稳定界面控制
对称的二节点网络能够维持极化界面的稳定(图1a)。然而,在单一节点上引入单边自我调控(图1b)、单边额外调控(图1c)、不对称系统参数(图1d),细胞极性斑图界面会持续往其中一侧持续入侵,以单一分子占据细胞膜全部区域终结。然而,当两种趋势相反的调控相结合时,界面的稳定性能够被恢复(图2)。
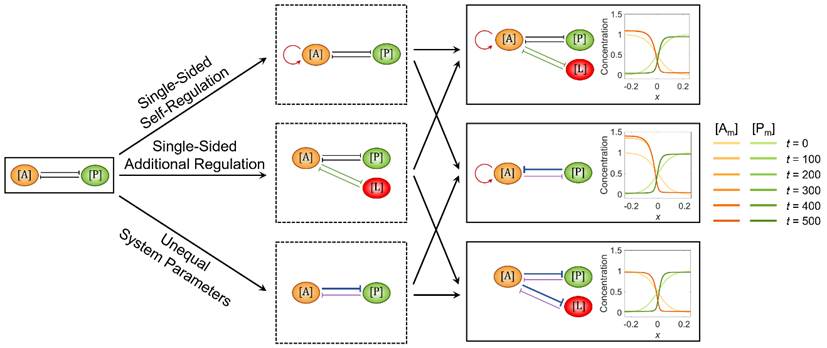
图2. 通过结合两种作用相反的单一调控,极性斑图可以被稳定维持。
从生物物理参数的定量上,当两种分子“势均力敌”时,界面将获得接近于零的速度,从而实现稳定的极性斑图。界面运动速度关于生物物理参数呈现三区域分离的相图,即向前崩塌、向后崩塌、或维持静止。具体如图3所示:案例1. 加入前部节点[A]的自激活(单边自我调控)及其与后部节点[L]的相互抑制(单边额外调控),可以获得自激活和相互抑制强度所构成的相图(图3a);案例2. 在原始的二节点网络结构上,6个生物物理参数彼此构成相图(图3b)。
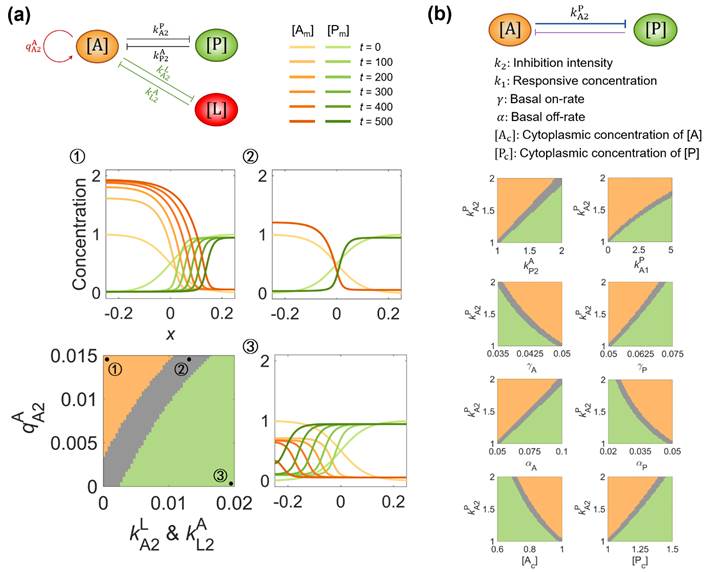
图3. 通过网络参数之间两两的相互权衡,斑图界面速度能够被精准控制,从而实现稳定细胞极性。
3. 现实线虫五节点网络的平衡设计
研究团队好奇上述网络平衡与界面控制是否在进化中已经被应用到自然生命体内。因此,研究团队调研了秀丽隐杆线虫受精卵极化维持过程中的完整分子网络;过去30年内该系统作为细胞极性研究的流行系统,已经报道了5个关键相互作用节点:PAR-3/PAR-6/PKC-3、CDC-42、PAR-1/PAR-2、LGL-1、CHIN-1(图4a) [Dev. Cell, 2015; Development, 2017; Dev. Cell, 2017]。该网络可被解构为一个翻倍的二节点拮抗网络(图4b),再加入前部节点[A]和[C]的相互促进(图4c)以及前部节点[A]和后部节点[L]的相互抑制(图4d)—— 该两侧网络结构修饰符合前述的平衡修饰结构之一(图3a;图4b, c, d)。除去网络结构设计以外,研究团队继续发现,野生型的五节点网络具有天然的进化优势,比四节点网络(或翻倍的二节点网络)具备在更大的可行生物物理参数空间和更强的参数鲁棒性(图4e, f, g)。
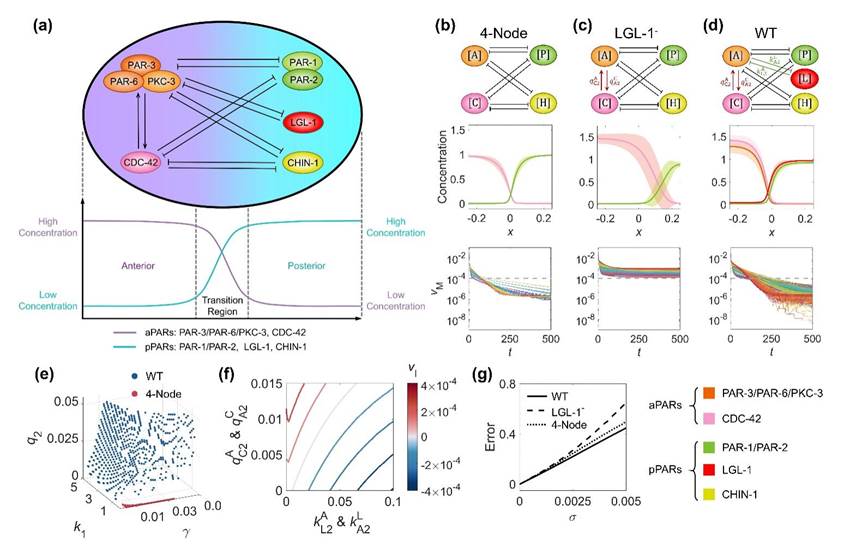
图4. 线虫受精卵极化的反应扩散网络及其在斑图稳定性、可行生物物理参数空间、参数鲁棒性等方面的进化优势。
随后,研究团队揭示了在高维空间下,界面运动速度可以由针对生物物理参数的线性展开获得(图5a);同时,具体的界面位置可以由多个参数的空间不对称分布锚定,对应现实生命体系中胞外信号等网络以外的影响因素 [Development, 2010; J. Theor. Biol., 2016](图5b-e)。上述两个定量调控策略为未来生物学家调控、合成细胞极性斑图提供了理论支持 [Cell, 2012; Cell, 2023]。
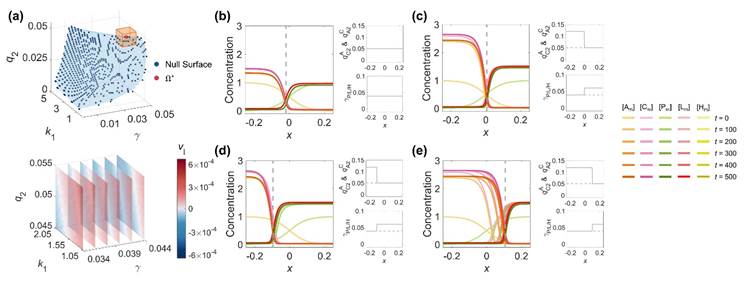
图5. 界面速度和位置可以通过生物物理参数进行预测和控制。
4. 开源软件
研究团队将本工作计算框架整合为开源软件PolarSim,支持其他实验和理论研究人员自定义网络构型、探索不同的生物物理参数对动力学行为的影响,可应用于不同生物体系的细胞极性模式研究(图6)。
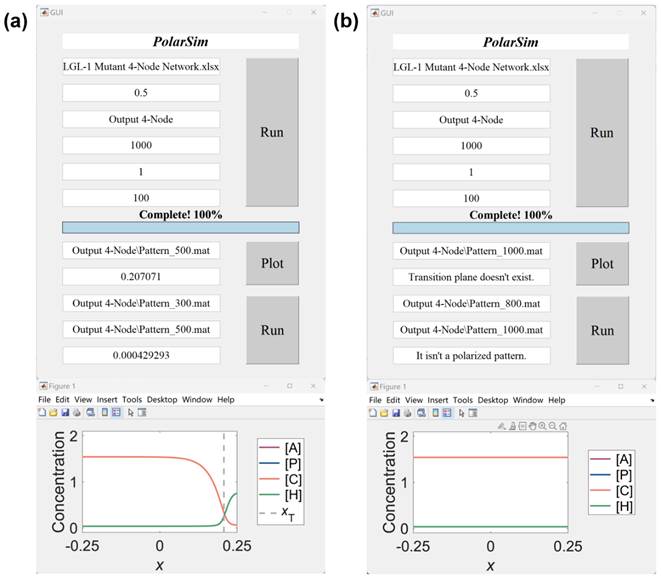

图6. PolarSim软件界面和输入输出。
5. 总结与展望
细胞极化在特定细胞位置构建起稳定的斑图界面,以实现空间信息读取和下游生理活动的正常执行。例如,秀丽隐杆线虫胚胎持续发生四轮细胞不对称分裂,其对子代细胞体积、命运的调控要求极化斑图高度精准且稳定 [Nat. Phys., 2019]。本研究团队近年围绕细胞不对称分裂的不同尺度进行了探究,包括定量数据采集与信号调控机制 [Nat. Commun., 2020; Nat. Commun., 2025]、细胞周期关联与发育速率优化 [Phys. Rev. E, 2019]、形态发生 [Phys. Biol., 2020; PLoS Comput. Biol., 2022]、跨物种保守性 [MicroPubl., 2024]。本工作为细胞不对称性的反应扩散网络与极性斑图形成,提供了新的物理视角。所报道的网络平衡原理与斑图控制机制可用于其他生物体系的网络分析,以及为未来人工合成具有细胞极化相关功能的生物学网络提供参考。本工作所搭建的线虫网络成功模拟前人报道的9组细胞极性实验现象,未来与其他尺度的模型、数据相结合,有望构建更加完备的多尺度发育模型。
北京大学定量生物学中心/生命科学联合中心/物理学院(现西湖大学交叉科学中心)汤超教授、松山湖材料实验室/香港浸会大学物理系/理论和系统研究所/环境与生物分析国家重点实验室(现西湖大学理学院及交叉科学中心)汤雷翰教授为文章的共同通讯作者;北京大学物理学院博士生陈宜煊(原四川大学物理学院本科生、松山湖材料实验室粤港澳交叉科学中心访问学生)、北京大学定量生物学中心博士生关国业(原松山湖材料实验室粤港澳交叉科学中心、香港浸会大学物理系访问学生;现哈佛医学院/丹娜法伯癌症研究院博士后)为文章的共同第一作者。北京大学高性能计算平台提供了计算支持。本工作由国家自然科学基金、香港研究资助局共同支持。
6. 文献
1) Goodrich LV, Strutt D. Principles of planar polarity in animal development. Development 2011;138:1877-92.
2) Etienne-Manneville S, Hall A. Rho GTPases in cell biology. Nature 2002;420:629-35.
3) Bilder D, Li M, Perrimon N. Cooperative regulation of cell polarity and growth by Drosophila tumor suppressors. Science 2000;289:113-6.
4) Macara IG. Parsing the polarity code. Nat Rev Mol Cell Biol 2004;5:220-31.
5) Kemphues KJ, Priess JR, Morton DG, et al. Identification of genes required for cytoplasmic localization in early C. elegans embryos. Cell 1988;52:311-20.
6) Cuenca AA, Schetter A, Aceto D, et al. Polarization of the C. elegans zygote proceeds via distinct establishment and maintenance phases. Development 2003;130:1255–65.
7) Tostevin F, Howard M. Modeling the establishment of PAR protein polarity in the one-cell C. elegans embryo. Biophys J 2008;95:4512-22.
8) Chau AH, Walter JM, Gerardin J, et al. Designing synthetic regulatory networks capable of self-organizing cell polarization. Cell 2012;151:320-32.
9) Kumfer KT, Cook SJ, Squirrell JM, et al. CGEF-1 and CHIN-1 regulate CDC-42 activity during asymmetric division in the Caenorhabditis elegans embryo. Mol Biol Cell 2010;21:266-77.
10) Beatty A, Morton D, Kemphues K. The C. elegans homolog of Drosophila lethal giant larvae functions redundantly with PAR-2 to maintain polarity in the early embryo. Development 2010;137:3995-4004.
11) Sailer A, Anneken A, Li Y, et al. Dynamic opposition of clustered proteins stabilizes cortical polarity in the C. elegans zygote. Dev Cell 2015;35:131-42.
12) Seirin-Lee S, Gaffney EA, Dawes AT. CDC-42 interactions with Par proteins are critical for proper patterning in polarization. Cells 2020;9:2036.
13) Lim YW, We FL, Shankar P, et al. A balance between antagonizing PAR proteins specifies the pattern of asymmetric and symmetric divisions in C. elegans embryogenesis. Cell Rep 2021;36:109326.
14) Lang CF, Munro E. The PAR proteins: From molecular circuits to dynamic self-stabilizing cell polarity. Development 2017;144:3405-16.
15) Rodriguez J, Peglion F, Martin J, et al. aPKC cycles between functionally distinct PAR protein assemblies to drive cell polarity. Dev Cell 2017; 42:400-15.e9.
16) Arata Y, Lee JY, Boldstein B, et al. Extracellular control of PAR protein localization during asymmetric cell division in the C. elegans embryo. Development 2010;137:3337-45.
17) Seirin-Lee S. Positioning of polarity formation by extracellular signaling during asymmetric cell division. J Theor Biol 2016;400:52-64.
18) Watson JL, Krüger LK, Ben-Sasson AJ, et al. Synthetic Par polarity induces cytoskeleton asymmetry in unpolarized mammalian cells. Cell 2023;186:4710-27.
19) Hubatsch L, Peglion F, Reich JD, et al. A cell-size threshold limits cell polarity and asymmetric division potential. Nat Phys 2019;15:1075-85.
20) Cao J, Guan G, Ho VWS, et al. Establishment of a morphological atlas of the Caenorhabditis elegans embryo using deep-learning-based 4D segmentation. Nat Commun 2020;11:6254.
21) Guan G, Li Z, Ma Y, et al. Cell lineage-resolved embryonic morphological map reveals signaling associated with cell fate and size asymmetry. Nat Commun 2025;16:3700.
22) Guan G, Wong M-K, Zhao Z, et al. Volume segregation programming in a nematode’s early embryogenesis. Phys Rev E 2021;104:054409.
23) Tian B, Guan G, Tang L-H, et al. Why and how the nematode's early embryogenesis can be precise and robust: A mechanical perspective. Phys Biol 2020;17:026001.
24) Kuang X, Guan G, Wong MK, et al. Computable early Caenorhabditis elegans embryo with a phase field model. PLoS Comput Biol 2022;18:e1009755.
25) Guan G, Luo C, Tang L-H, et al. Modulating cell proliferation by asymmetric division: A conserved pattern in the early embryogenesis of nematode species. MicroPubl Biol 2024;10.17912/micropub.biology.001006.