小分子药物研发的成本高、周期长,并且有着很高的失败风险。全新分子设计(De novo molecule design)方法能够根据已有的信息自动探索化学空间,以期发现潜在的高活性、高类药性分子结构。北京大学化学与分子工程学院/前沿交叉学科研究院来鲁华、裴剑锋团队开发的LigBuilder程序[1-5]就是一种具有代表性的全新分子设计方法,该程序已经成功应用于诸多靶标的抑制剂设计工作(包括亲环素 A、ABL 激酶、IκB kinase β等)。近年来,随着人工智能和深度学习技术的发展,深度分子生成模型(Deep molecule generative model)作为一种新兴的全新分子设计方法为该领域注入了新的活力。相比传统方法,基于深度学习的模型能够实现基于数据的自动化训练,无需专家知识的集成,并且具有很强的表现力和泛化能力。
现有的深度分子生成模型大多数只能生成分子的二维拓扑结构,在基于已知配体的药物设计任务(Ligand-based drug design,LBDD)中表现良好。但由于这些方法尚无法直接设计分子的三维(3D)结构,因此难以实现基于结构的药物设计(Structure-based drug design,SBDD),即利用靶标蛋白口袋的结构信息设计分子三维结构。SBDD在药物设计中有着重要的地位,配体和受体在三维空间中的相互作用信息能够为药物设计提供更直接的指导,在没有已知活性分子可以参考的情况下也可以直接设计与靶标结合的分子。发展集成深度分子生成模型和SBDD的方法将为全新药物设计带来新机遇。
近期,来鲁华、裴剑锋团队继续发展了一种基于深度学习的全新3D分子设计方法[MAIN],能够直接在靶标口袋内生成与之结合的三维分子结构。该工作的初稿于2021年4月以预印本的形式发表在arXiv平台上(https://arxiv.org/abs/2104.08474),9月21日该工作论文以“ Structure-based de novo drug design using 3D deep generative models”为题正式发表于Chemical Science杂志(https://pubs.rsc.org/en/content/articlelanding/2021/SC/D1SC04444C)。该论文还选入2021 Chemical Science HOT Article Collection。来鲁华教授和裴剑锋研究员为论文的通讯作者,论文的第一作者为北京大学前沿交叉学科研究院生命科学联合中心的博士研究生李亦博。
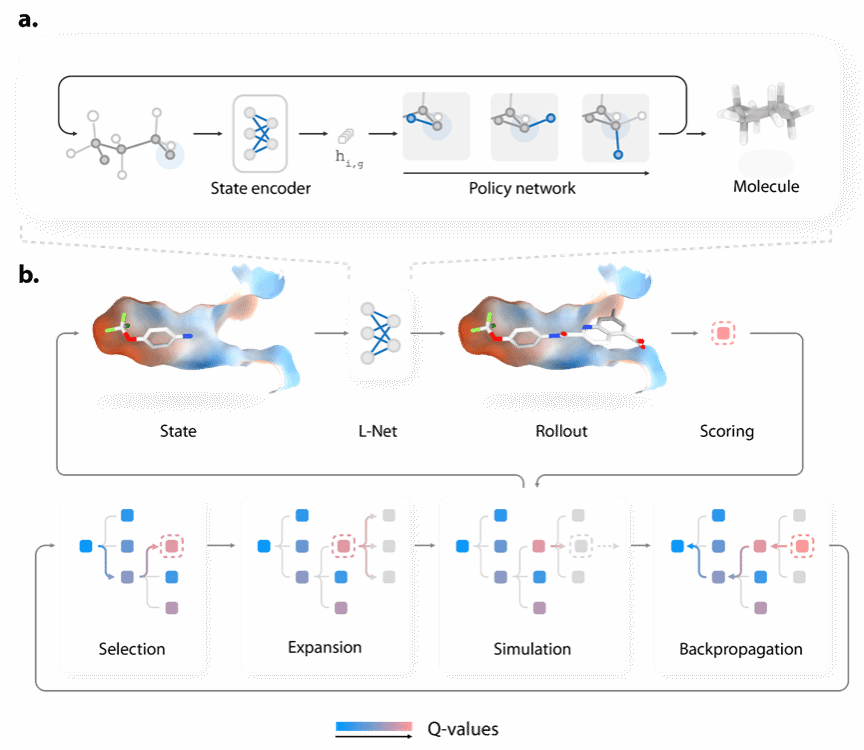
图1:DeepLigBuilder的模型构架。DeepLigBuilder由两部分组成:(a)能够实现端到端3D类药分子生成的L-Net;(b)负责在靶标口袋中优化3D分子结构的MCTS算法。
为了实现基于结构的三维分子设计,该团队首先开发了一种全新的网络构架—配体神经网络(Ligand neural network,L-Net)—以实现端到端的三维分子构建。L-Net基于图卷积神经网络,利用从ChEMBL数据库中提取的分子结构进行训练,能够生成包含高质量3D构象的类药分子。接下来,该团队将L-Net与MCTS(Monte Carlo tree search)算法相结合实现了基于靶标结构的三维分子设计,并开发了相应的软件DeepLigBuilder。以SARS-CoV-2主蛋白酶的抑制剂设计作为案例,DeepLigBuilder能够生成具有高类药性、高可合成行以及高预测结合活性的分子,所设计的分子能够很好地再现已有阳性分子的结合模式,同时也生成了新颖的化学骨架。
基于结构的三维分子生成是未来AI药物设计研究的重要新方向。DeepLigBuilder首次将3D图生成网络用于SBDD,也是首个利用深度学习和MCTS优化三维分子结构的工作。在未来工作中,该团队计划对DeepLigBuilder生成的分子进行活性验证,并将DeepLigBuilder的应用场景拓展到其他领域,如功能小分子的设计中。该工作主要得到了国家自然科学基金重点项目(22033001)和重大新药创制科技重大专项(2018ZX09711002)的支持。
参考文献:
[MAIN] Li, Yibo, et al. “Structure-Based de Novo Drug Design Using 3D Deep Generative Models.” Chemical Science, 2021, doi:10.1039/d1sc04444c.
[1] “LigBuilder V3 - An Integrated De Novo Drug Design System”, http://www.pkumdl.cn/ligbuilder/
[2] Yuan, Yaxia, et al. “LigBuilder V3: A Multi-Target de Novo Drug Design Approach.” Frontiers in Chemistry, vol. 8, 2020, p. 142, doi:10.3389/fchem.2020.00142.
[3] Qing, Xiaoyu, et al. “Multiple Target Drug Design Using LigBuilder 3” Methods in Molecular Biology, vol. 2266, 2021, pp. 279–98, doi:10.1007/978-1-0716-1209-5_16.
[4] Yuan, Yaxia, et al. “LigBuilder 2: A Practical de Novo Drug Design Approach.” Journal of Chemical Information and Modeling, vol. 51, no. 5, 2011, pp. 1083–91, doi:10.1021/ci100350u.
[5] Wang, Renxiao, et al. “LigBuilder: A Multi-Purpose Program for Structure-Based Drug Design.” Molecular Modeling Annual, vol. 6, no. 7–8, 2000, pp. 498–516, doi:10.1007/s0089400060498.