在真核细胞中,蛋白质与核酸等生物大分子通过相分离形成生物分子凝聚物,在基因表达调控、胞内信号传导等重要生命活动中发挥精准的时空调控作用 1。近年来,越来越多具有特殊结构的凝聚物被报道,如“珍珠链”结构、“俄罗斯套娃”式嵌套结构、内部具有生物分子低密度腔室的空洞结构等 2-5。这些不同的结构对应独特的形成机制与生物学功能。其中,球壳凝聚物作为结构最为特殊的凝聚物类型,其形成机制一直是生物物理领域的研究重点与难点。
近日,北京大学定量生物学中心齐志课题组、张磊课题组、钱珑课题组与中国科学院过程工程研究所夏宇飞课题组在FET融合致癌蛋白形成球壳凝聚物的研究取得突破性进展。他们发现FET家族融合致癌蛋白FUS-ERG可与含GGAA微卫星序列的双链DNA(dsDNA)形成独特的球壳共凝聚物,并通过体外生化实验、超分辨成像与数学建模相结合的方法,揭示了其“嵌套不对称相分离”的形成机制”。更具独创性的是,研究人员基于FUS-ERG-dsDNA球壳共凝聚物的结构特性,研究人员开发出独特的DNA操控技术,成功实现DNA信息的精准删除及层级分选,为DNA存储领域提供了全新技术路径。相关论文“Deciphering the molecular mechanisms of FET fusion oncoprotein–DNA hollow co-condensates”已发表于Nature Communications。

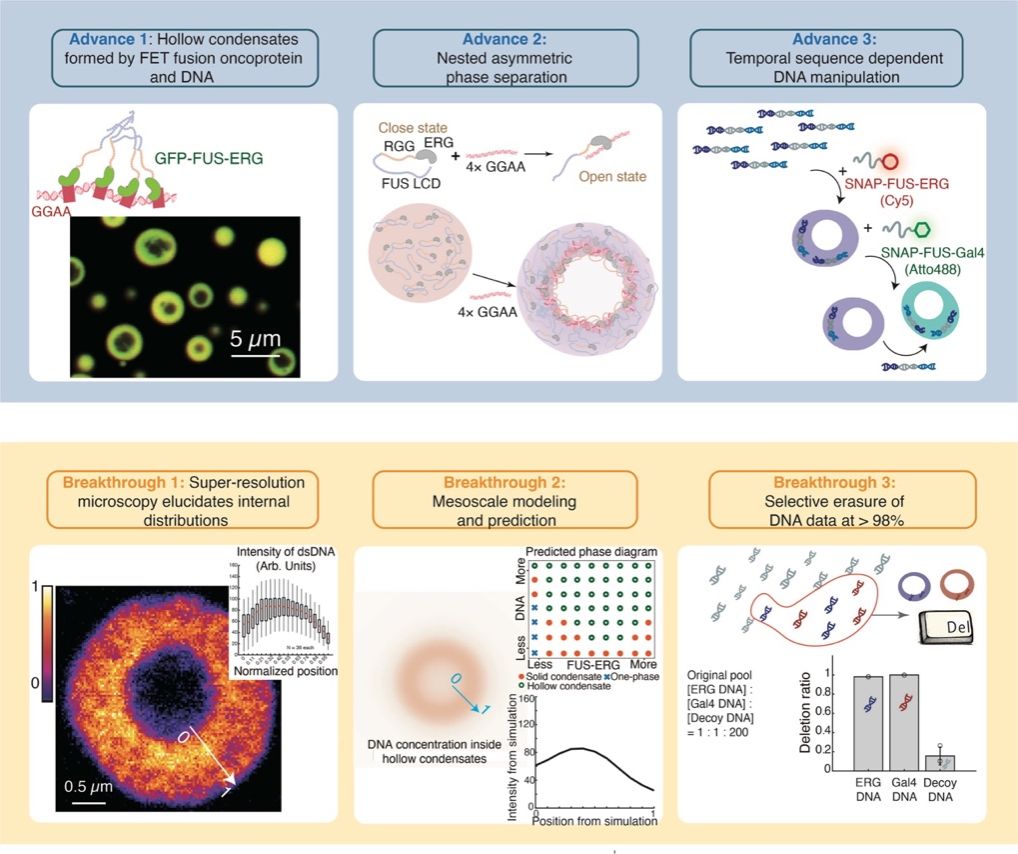
FUS-ERG是FUS/EWS/TAF15(FET)家族中一种典型的融合致癌蛋白,由FUS的低复杂度结构域(LCD)、RGG结构域与ETS家族转录因子ERG的DNA结合结构域(DBD)融合而成,可特异性识别基因组中的GGAA微卫星重复序列。体外液滴实验发现,FUS-ERG可以与随机序列的25-bp dsDNA形成内部均质的液滴型凝聚物;但当dsDNA中存在GGAA重复序列时,两者可自发组装成球壳共凝聚物,蛋白与dsDNA共同富集于球壳结构的壳层区域(图1)。将FUS-ERG蛋白LCD中的酪氨酸(Y)突变为丝氨酸(S),或将RGG中的精氨酸(R)的突变为丙氨酸(A)均会导致蛋白无法和GGAA dsDNA形成球壳共凝聚物。证明蛋白与GGAA序列的特异性结合、蛋白结构域之间的相互作用是球壳共凝聚物形成的关键要素。
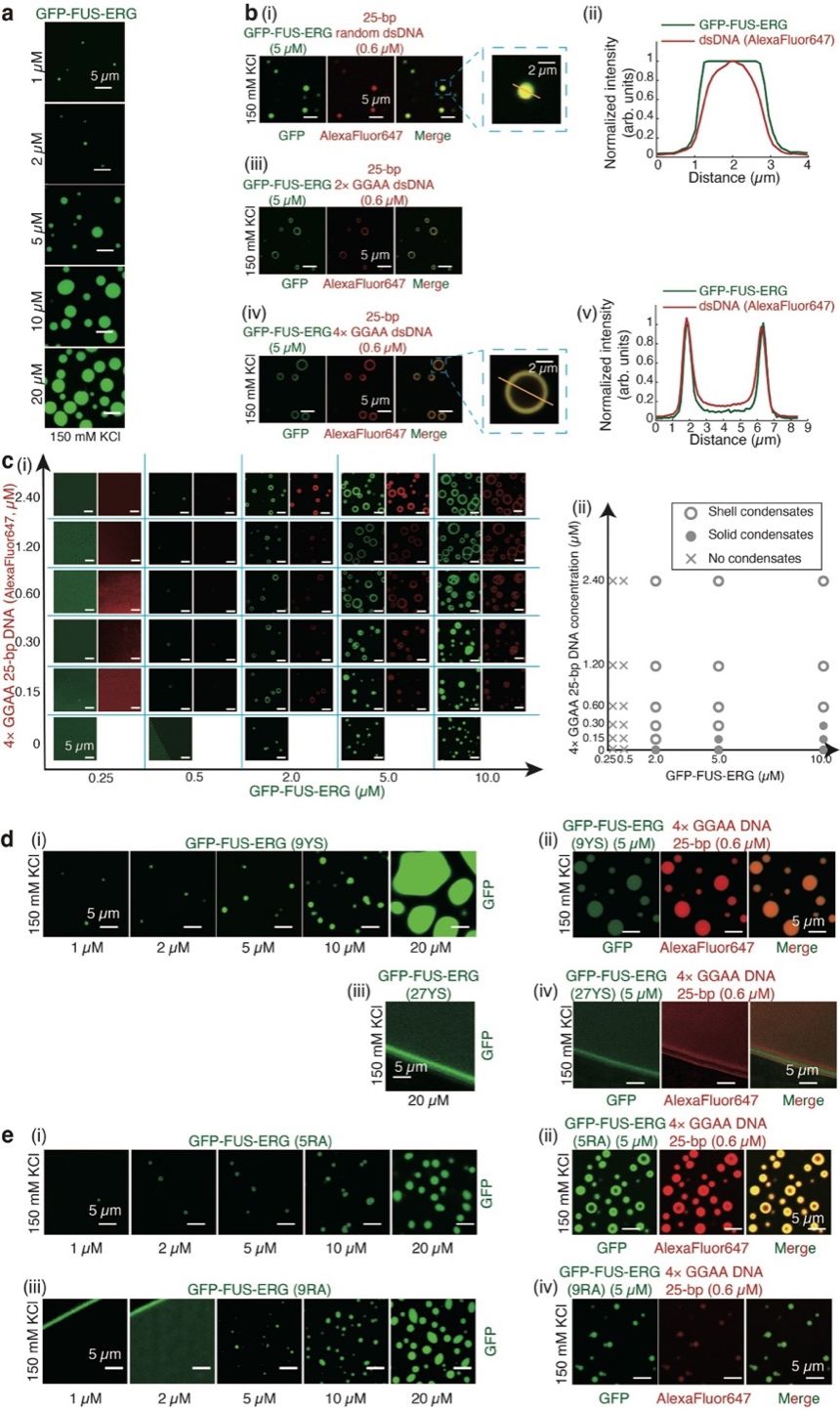
图1. FUS-ERG与GGAA dsDNA形成球壳共凝聚物。
球壳型凝聚物具有一层特殊的内表面。研究团队通过向体系中加入RNA探针,探究FUS-ERG-dsDNA球壳共凝聚物内表面的生物物理性质,研究团队向体系中加入了RNA探针,发现RNA能高效穿透壳层并富集在腔体内,但无法渗透进入实心均质的凝聚物中(图2)。由于RGG结构域是FUS-ERG中唯一能有效结合RNA的区域,这一结果提示球壳共凝聚物的内表面暴露了大量RGG基序,而在实心均质的凝聚物中RGG基序则可能被埋藏在蛋白内部。
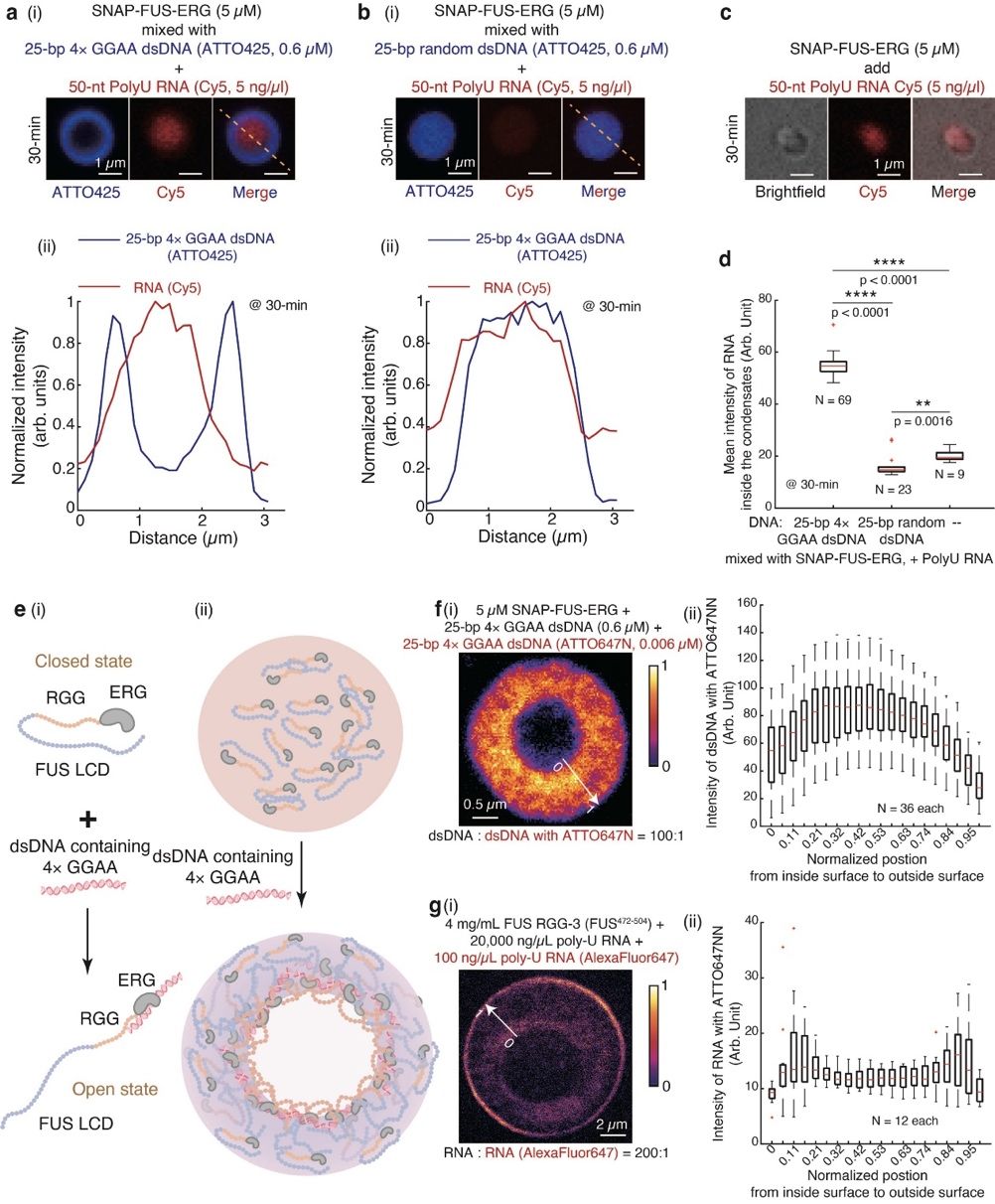
图2. 嵌套不对称相分离驱动FUS-ERG-dsDNA球壳共凝聚物的形成
为此,研究团队提出“嵌套不对称相分离”假说:FUS-ERG存在“闭合态”与“开放态”两种构象。在闭合态中,LCD与ERG结构域通过分子间相互作用紧密结合,RGG基序被埋藏;当GGAA dsDNA特异性地结合到ERG的DBD后,触发构象转变为开放态,RGG基序得以暴露。这些开放态的蛋白-DNA复合物向凝聚物内部扩散,暴露的亲水性RGG基序在内部积累形成亲水性内表面,而外部表面则由闭合态蛋白与缓冲液的相互作用维持稳定,最终形成内外表面不对称的中空结构(图2)。
研究团队验证了这两种构象的合理性:使用体外生化实验观测到LCD与ERG结构域之间的相互作用,发现含有含GGAA序列的dsDNA能稳定其开放态构象。为确认蛋白-DNA复合物可以向凝聚物内部扩散并聚集,团队使用受激发射损耗显微镜(STED)超分辨成像发现球壳凝聚物壳层上的dsDNA荧光强度呈现“内高外低”的径向分布特征(图2),证实了蛋白-DNA复合物可以向凝聚物内部扩散并聚集,与团队预测假说高度吻合。与此同时,在对照组鱼精蛋白PRM-RNA囊泡状球壳凝聚物的壳层上,RNA的分布高度对称,说明两种球壳凝聚物的形成机制显著不同 5。
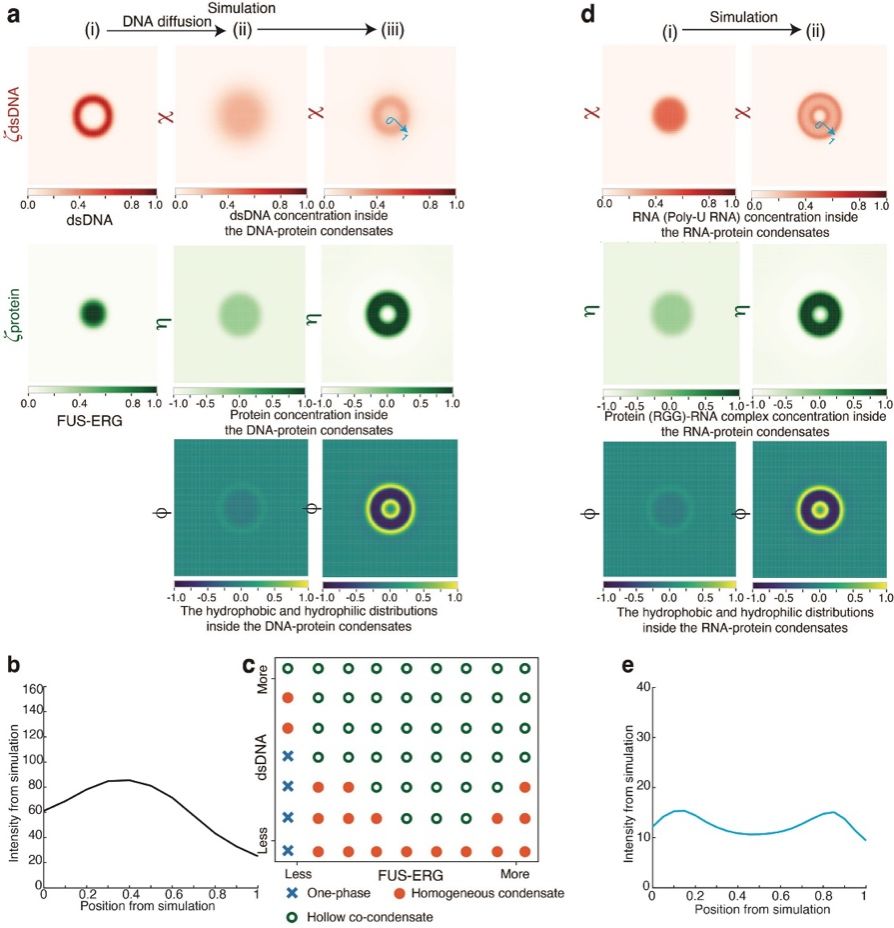
图3. 介观尺度的分子信息相场模型可复现并区分两种球壳凝聚物的形成机制
为从物理机制层面定量解析球壳共凝聚物的形成过程,团队开发了一个整合了分子信息的介观尺度相场模型。该模型巧妙地将蛋白-DNA复合物的浓度、疏水/亲水特性等关键分子信息转化为数学语言,成功模拟了从初始的DNA表面吸附到最终形成稳定球壳结构的完整动态过程。该模型不仅精确复现了球壳结构的形成,其预测的dsDNA在壳层上“内高外低”的不对称分布也与STED超分辨成像数据定量一致。同时,通过调整代表“结合亲和力”或“疏水性”的参数,模型能够分别再现使用低亲和力DNA或蛋白突变体时无法形成球壳结构的实验结果,从理论上验证了“嵌套不对称相分离”假说的物理基础。此外,该模型还成功区分了本研究中发现的“不对称”球壳结构与文献报道的“对称”囊泡结构,并成功预测了凝聚物对不同亲和力DNA的选择性排斥行为,该预测也为后续实验所证实,充分展示了模型的解释力与预测能力(图3)。
此外,研究团队还构建了具有不同DBD的FET融合蛋白,例如把FUS-ERG中ERG DBD替换为Gal4 DBD得到FUS-Gal4融合蛋白,该人造的融合蛋白只能和具有Gal4特异结合序列的UAS dsDNA形成球壳共凝聚物,而FUS-ERG只能和GGAA dsDNA形成球壳凝聚物一致(图4)。这不仅证明了FET融合蛋白与其对应dsDNA形成球壳凝聚物的分子机制是高度通用的,还提供了更多正交的反应体系,对于解决当前DNA存储领域的技术瓶颈提供有力的技术支持。
在DNA存储中,数据被编码到短DNA文库中,每个短DNA片段都带有独特的条形码序列,为后期的选择性随机访问提供索引。当前DNA存储领域面临的核心挑战之一是信息分子的特异性删除与动态调控,传统的磁珠技术操作步骤繁琐,并且容易造成非靶标DNA片段的损失,难以满足高密度存储文库的动态管理需求的缺陷。研究团队借助FUS-Gal4和FUS-ERG仅识别各自特异DNA序列的特性,研究团队将FUS-Gal4和FUS-ERG两种蛋白投入至混合了大量随机序列的DNA文库中,发现UAS dsDNA和GGAA dsDNA的删除效率接近100%,随机序列DNA的删除率仅为约20%;与之相对,无法形成球壳结构的9YS突变体融合蛋白虽然也能删除目标序列,但对随机序列DNA的删除率高达70%(图4)。这一结果证实球壳结构是实现高特异性删除的关键,可有效减少非特异删除造成的信息损失。更振奋的是,团队实现了DNA存储的动态层级数据选择:将GGAA和UAS序列作为多级信息单元条形码,先加入FUS-ERG形成球壳凝聚物,实现第一层级信息选择;之后向体系中加入FUS-Gal4,FUS-Gal4可完全剔除FUS-ERG并形成新的球壳凝聚物(图4),实现对DNA文件的第二层级选择。
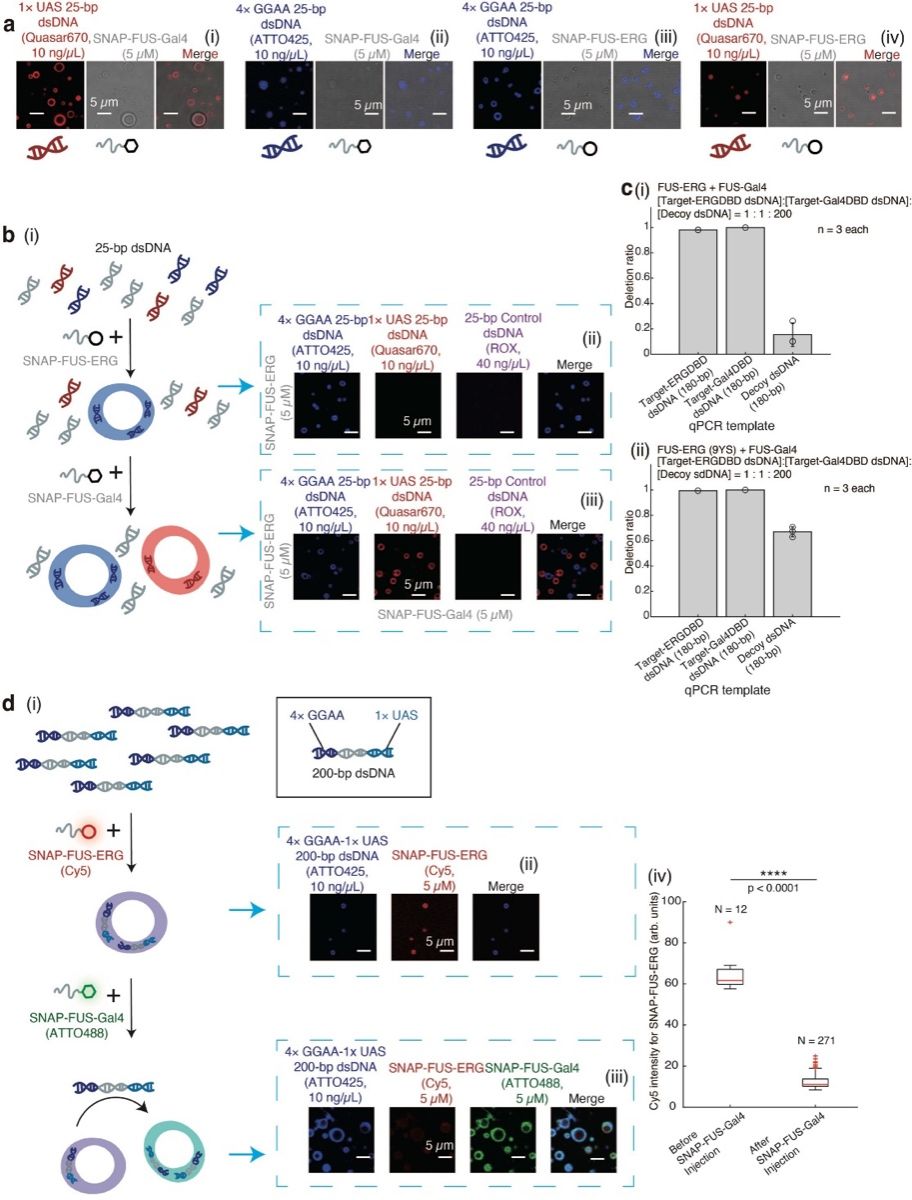
图4. 基于FET融合蛋白-dsDNA球壳共凝聚物的DNA信息操控应用
该研究通过生物化学、生物物理、数学建模与信息科学的深度交叉,实现了基础理论与应用技术层面的双重突破。在基础研究领域,首次阐明了FET融合致癌蛋白与dsDNA形成球壳共凝聚物的“嵌套不对称相分离”机制,为理解多组分凝聚物的结构多样性提供了新的理论框架,为相关疾病的治疗提供了全新思路。在应用技术领域,基于球壳共凝聚物开发的DNA操控技术具有显著优势:一是“微创”操作,能将对非目标DNA文库的破坏控制到较低水平,最大限度地保持了文库完整性;二是“可编程”识别,通过替换不同DBD可实现对任意目标序列的特异性识别;三是“动态”调控,利用不同DBD-DNA的结合实现信息单元的原位动态切换。这些特性使其在DNA存储、分子计算等领域展现出巨大的应用潜力。
北京大学定量生物学中心齐志教授、张磊教授、钱珑副研究员、中国科学院过程工程研究所夏宇飞研究员为本文共同通讯作者。北京大学定量生物学中心博士后左琳彧、博士生郭骐瑞、博士后李承为本文共同第一作者。北大-清华生命科学联合中心张珂诚博士、北京大学定量生物学中心博士后陈岩草、博士生姜百翼、北大-清华生命科学联合中心陈知行研究员为本研究做出重要贡献。本研究获得了国家重点研发计划“生物与信息融合(BT与IT融合)”重点专项(2023YFF1205600),和自然科学基金委杰青基金(T2225009 to Z.Q.)等项目支持。
文章链接:https://www.nature.com/articles/s41467-025-65069-4
齐志课题组(北京大学前沿交叉学科研究院定量生物学中心):聚焦研发新型多维度、跨尺度单分子技术,并把非平衡态物理学融入交叉研究,解析生物大分子动态聚集体的分子机理:1. 转录调控中的核酸-蛋白共凝聚物的分子机制;2. DNA修复中的单链DNA-RPA复合物的分子机制;3. RNA biology中的RNA-蛋白共凝聚物的分子机制;欢迎已获得或短期内可获得物理、化学、生物、或工程等相关专业博士学位的有志科研之士申请博士后及科研助理职位。按照北京大学相关规定执行,提供具有国际竞争力的待遇条件,优秀者推荐申请“北大博雅博士后基金”,具体待遇面议。申请者请将个人简历投递zhiqi7@pku.edu.cn(应聘理由请注明“博士后申请+姓名”)。本招聘长期有效。导师信息及相关个人页面:http://cqb.pku.edu.cn/qizhi/ (联系方式:zhiqi7@pku.edu.cn)。
张磊课题组(北京大学北京国际数学研究中心、定量生物学中心、国际机器学习中心):研究领域为计算与应用数学、数学与生命科学交叉,包括非线性复杂系统的“解景观”方法与应用;复杂生物系统的可计算建模、生物网络的多功能设计原理、细胞命运与发育轨迹的多组学数据推断、植物形态建模;科学计算与机器学习等。欢迎已获得或短期内可获得数学、定量与系统生物学、数据科学与人工智能等相关专业博士学位的有志科研之士申请博士后及助理/副科研员职位,将提供具有国际竞争力的待遇条件,本招聘长期有效。导师信息及相关个人页面:http://bicmr.pku.edu.cn/~zhanglei (联系方式:pkuzhangl@pku.edu.cn)
钱珑课题组(北京大学前沿交叉学科研究院定量生物学中心):从事生物信息、生物合成和生物工程的交叉研究。综合运用深度学习、生物信息学、合成生物学和单分子测序等技术手段,聚焦DNA分子信息存储、基因元件的大数据挖掘与设计、合成基因线路的可预测设计、基因组系统进化等问题。欢迎生物学、计算科学、数理科学等相关专业学者申请博士后及科研助理职位。按照北京大学相关规定执行,提供具有国际竞争力的待遇条件。申请者请将个人简历投递long.qian@pku.edu.cn。本招聘长期有效。个人主页:https://cqb.pku.edu.cn/info/1002/1812.htm,实验室主页: https://www.bdainformatics.org/。
夏宇飞课题组(中国科学院过程工程研究所):聚焦疫苗黏膜佐剂、肿瘤疫苗、mRNA递送系统及其组装和递送微观过程研究。欢迎已获得或短期内可获得化学、生物、或工程等相关专业博士学位的有志科研之士申请博士后及科研助理职位。按照中国科学院过程工程研究所相关规定执行,提供具有国际竞争力的待遇条件,优秀者推荐申请“特别研究助理”计划,具体待遇面议。申请者请将个人简历投递yfxia@ipe.ac.cn。本招聘长期有效。导师信息及相关个人页面:
https://people.ucas.edu.cn/~yufeixia?language=en。
文献
1 Banani, S. F., Lee, H. O., Hyman, A. A. & Rosen, M. K. Biomolecular condensates: organizers of cellular biochemistry. Nat Rev Mol Cell Bio 18, 285-298, doi:10.1038/nrm.2017.7 (2017).
2 Li, C. et al. Deciphering the molecular mechanism underlying morphology transition in two-component DNA-protein cophase separation. Structure 33, 62-77.e68, doi:10.1016/j.str.2024.10.026 (2025).
3 Feric, M. et al. Coexisting Liquid Phases Underlie Nucleolar Subcompartments. Cell 165, 1686-1697, doi:10.1016/j.cell.2016.04.047 (2016).
4 Banerjee, P. R., Milin, A. N., Moosa, M. M., Onuchic, P. L. & Deniz, A. A. Reentrant Phase Transition Drives Dynamic Substructure Formation in Ribonucleoprotein Droplets. Angew Chem Int Edit 56, 11354-11359, doi:10.1002/anie.201703191 (2017).
5 Alshareedah, I., Moosa, M. M., Raju, M., Potoyan, D. A. & Banerjee, P. R. Phase transition of RNA-protein complexes into ordered hollow condensates. P Natl Acad Sci USA 117, 15650-15658, doi:10.1073/pnas.1922365117 (2020).